2019 Volume 68 Issue 3 Pages 455-462
2019 Volume 68 Issue 3 Pages 455-462
固定における種類や時間に関する推奨法はガイドライン等で明記されているが,脱灰液に関する詳細な記述はほとんどみられない。今回,われわれは,遺伝子検索における脱灰の影響について検討を行った。方法は,種々の脱灰液および脱灰時間の標本を用いて,DNA収量およびポリメラーゼ連鎖反応(polymerase chain reaction; PCR)増幅について検討した。また,既知の上皮成長因子受容体(human epidermal growth factor receptor; EGFR)変異陽性と変異陰性1症例を対象についても検索した。結果は,強酸系脱灰液では,DNA収量は少なく,PCR法による増幅は確認できなかった。10%ギ酸ホルマリンでは,脱灰時間が長くなるほどDNA収量は減少した。一方,エチレンジアミン四酢酸(ethylene diaminetetraacetic acid; EDTA)ではDNA収量が多く,PCR増幅も100 bp~400 bpまで良好な結果が得られた。脱灰時間に関しても7日間処理を行ってもDNA収量は減少せず良好な結果であった。また,肺腺癌におけるEGFR変異検出キットにおいては,EDTA 7日間処理でも検出可能であった。遺伝子変異解析に用いる組織標本において脱灰処理が必要な場合,EDTAを脱灰液として用いることが肝要である。
In recent years, gene search from pathological specimens has been indispensable for the development of molecular targeted therapeutic drugs, and the results determine the suitability of therapeutic agents. Recommendations on the type and time of specimen fixation are specified in guidelines and protocols, but detailed descriptions concerning decalcification agents are rarely seen. In this study, we examined the effect of decalcification on gene search. The DNA yield and PCR amplification were compared among FFPE specimens prepared using various decalcification agents and decalcification times. We also determined the effects of various decalcification agents on EGFR mutation. The results showed that DNA yield was low in the strong-acid-type decalcification agents K-CX and Plank-Rychlo, and no amplification by PCR was observed. Also, with the strong-acid-type decalcification and 10% formalin formic acid, the DNA yield decreased as the decalcification time increased. On the other hand, 10% EDTA yielded more DNA, and PCR amplification was good from 100 to 400 bp. Regarding decalcification time, DNA yield did not decrease even after 7 days of treatment, which was a good result. In the study of the EGFR mutation detection kit for pulmonary adenocarcinoma, detection with strong-acid-type decalcification agents and 10% formalin formic acid was impossible regardless of decalcification time, but EGFR mutation can be detected with 10% EDTA treatment for 7 days. When decalcification is required for mutation search, it is essential to use EDTA as a decalcification agent.
近年,分子標的治療薬の開発に伴い病理標本からの遺伝子検索は必須であり,その結果は治療薬剤の適否を決定する。現在では,ホルマリン固定パラフィン包埋(formalin fixed paraffin embedded; FFPE)標本から,上皮成長因子受容体(human epidermal growth factor receptor-2; HER2)遺伝子増幅,上皮成長因子受容体(human epidermal growth factor receptor; EGFR)遺伝子変異,RAS遺伝子変異など種々の遺伝子検索が行われるようになった。対象となる臓器や遺伝子別にガイドラインや手引き書が作成され,検体の取り扱いについての重要性や精度管理について記載されている1),2)。しかしながら,固定液の種類や時間などについては明記され推奨法が指定されているが,脱灰処理に関する詳細な記載はほとんどない。脱灰に関しては,日本病理学会ゲノム研究用病理組織検体取扱い規定に急速脱灰(プランク・リュクロ法)を回避し,エチレンジアミン四酢酸(ethylene diaminetetraacetic acid; EDTA)による緩徐脱灰を行うべきであると記載されており,1症例の実証データが示されているのみである。遺伝子検索は,用いる脱灰液によっては,検索困難な場合があるが,その詳細な報告は少ない。
そこで今回,われわれは,遺伝子検索における脱灰の影響について検討を行った。種々の脱灰液のFFPE標本を用いて,DNA収量およびポリメラーゼ連鎖反応(polymerase chain reaction; PCR)増幅について比較検討し,また,既知のEGFR変異陽性1症例と変異陰性1症例を対象に,体外診断用医薬品承認されているEGFR変異検出キットにおける各種脱灰液の影響について検討した。
インフォームドコンセントで検体の使用を認められた肺腺癌と大腸癌の各5症例を対象とした。方法は10%ホルマリンにて12~24時間固定した組織を1 cm大,厚さ3 mmに分割し,脱灰液処理無し(コントロール),10%ギ酸ホルマリン,10% EDTA2Na pH 7.0中性脱灰液(以下EDTA,武藤化学株式会社)と塩酸をベースとしたK-CX®︎(株式会社ファルマ),プランク・リュクロ脱灰液(武藤化学株式会社)の強酸系脱灰液にそれぞれ,1日,3日,7日間処理した後,自動包埋装置(ティーシュー・テックVIP6®:サクラファインテックジャパン)にてFFPEブロックを作製した。
各ブロックについて4 μm切片のヘマトキシリン・エオジン(Hematoxylin-Eosin; HE)染色標本を作製し,各種脱灰液のHE染色標本に対する影響について観察した。
DNA抽出には,10 μm切片を10枚作製後スライドガラスに貼り付け,脱パラフィン後,切片をカミソリにて削り取りマイクロチューブに回収した。DNA抽出キットは,QIAamp DNA FFPE Tissue Kit®(キアゲン社)を用い指定のプロトコールに則り抽出を行った。DNA収量の測定には,Qubit® 2.0 Fluorometer(サーモフィッシャーサイエンティフィックス社)を用いて,DNA濃度測定を行った(Table 1)。PCR法はBIOMED-2 control primer set3)のうち100~400 bpのprimerをmixしPCR反応を行った(Table 2)。
| [DNA抽出] |
|---|
| 1.脱パラフィン:Xylene 10分 × 3回,100% Ethanol 10分 × 3回 |
| 2.風乾 |
| 3.180 μLのATL bufferにProteinase K 20 μLを加える |
| 4.カミソリで検体を削りチューブ内へ入れ Vortex 15秒混和しspin downする |
| 5.56℃で加温し1晩反応 |
| 6.90℃で加温し1時間反応 |
| 7.AL buffer 200 μLを加えVortex 15秒混和しspin downする |
| 8.Ethanolを加え200 μL Vortex 15秒混和しspin downする |
| 9.室温5分放置 |
| 10.sampleをカラムへ移し室温2分放置 |
| 11.20℃ 8,000回転 1分 遠心 |
| 12.AW1 buffer 500 μLを加え室温2分放置 |
| 13.20℃ 8,000回転 1分 遠心 |
| 14.AW2 buffer 500 μLを加え室温2分放置 |
| 15.20℃ 8,000回転 1分 遠心 |
| 16.新しいチューブへ交換後,14,000回転 1分 遠心 |
| 17.カラムを1.5 mLチューブへ移し,20 μL ATE bufferを加える |
| 18.室温5分放置 |
| 19.20℃ 14,000回転 1分 遠心後冷蔵保存 |
| [DNA濃度測定] |
| Qubit® ds DNA BR Assay Kitを用いQubit® 2.0 Fluorometerにて測定 |
QIAamp DNA FFPE Tissue KitにおけるプロトコールとQubit® 2.0 Fluorometerでの濃度測定キットを示す。
| 1.PCR反応液の調整(20 μL) |
|---|
| 各サイズのPrimerは,TBXAS(100 bp),RAG1(200 bp),PL2F(300 bp),AF4(400 bp)を使用した。 |
| PCR反応溶液の組成は,Ampli Taq Gold® 360 Master Mix:10 μL,10 μM Forward-Primer 0.3 μL,10 μM Reverse-Primer:0.3 μL,使用DNA量200~300 ngになるように調整(最大量7.6 μL使用)し,全反応溶液量を20 μLとした。 |
| 2.PCR反応 |
| GeneAmp® PCR System 9700(Applied Biosystems)を用い,95.0℃ 15分加熱後,95.0℃ 45秒,60.0℃ 45秒,72.0℃ 90秒のサイクルを35繰り返し,72.0℃で10分で加熱した後16.0℃とした。 |
| 3.電気泳動 |
| PCR産物 μLを1X TAE Bufferで作製した2%アガロースゲルでMupid-2 plus®(ADVANCE)1ooVにて30分電気泳動を実施した。泳動後,エチジウムブロマイドで30分染色し,その後ゲル撮影装置でDNAバンドを撮影した。 |
PCR sample調整の試薬量とPCR反応条件および電気泳動条件を示す。
また,肺腺癌症例に関しては,既知のEGFR変異陽性症例と陰性症例の2症例を対象に,体外診断用医薬品承認されているコバス® EGFR変異検出キットと核酸増幅・検出装置「コバス4800システム z480」を用い検索を行った。
肺腺癌(L1)と大腸癌(C1)のHE染色標本における各種脱灰液別の経時的変化をFigure 1, 2に示す。10%ギ酸ホルマリンおよびEDTAでは,7日間まではHE染色結果に影響が少なく核や細胞質の染色性が比較的良好に保たれていた。それに比べK-CXやプランク・リュクロ脱灰液では,3日経過ごろからヘマトキシリン(核)の染色性が低下し,エオジン(細胞質)の染色性が増強されており,いわゆる好酸化の目立つ染色結果であった。

10%ギ酸ホルマリンおよびEDTAでは,7日間まではHE染色結果に影響が少なく核や細胞質の染色性が比較的良好に保たれていた。それに比べK-CXやプランク・リュクロ脱灰液では,3日経過ごろからヘマトキシリン(核)の染色性が低下し,エオジン(細胞質)の染色性が増強されておりいわゆる好酸化の目立つ染色結果であった。

肺腺癌と同様の染色結果である。形態観察からのDNAの断片化の判断は難しい。
肺腺癌症例L1~L5,大腸癌症例C1~C5におけるDNA収量濃度をTable 3, 4と肺腺癌における収量における比較をFigure 3のグラフに示す。
| L1 | L2 | L3 | L4 | L5 | ||
|---|---|---|---|---|---|---|
| 1日間 | 処理なし | 986 | 268 | 434 | 436 | 600 |
| 10%ギ酸 | 149(15.1) | 113(42.2) | 63.2(14.6) | 37.6(8.6) | 148(24.7) | |
| EDTA | 838(84.5) | 528(197) | 454(105) | 510(169) | 390(65) | |
| K-CX | 12.4(1.3) | < 0.010(< 0.01) | < 0.010(< 0.01) | 4.24(1.0) | < 0.010(< 0.01) | |
| P-L | 12.4(1.3) | < 0.010(< 0.01) | < 0.010(< 0.01) | 4.16(1.0) | < 0.010(< 0.01) | |
| 3日間 | 10%ギ酸 | 31.4(3.2) | 7.16(2.7) | 9.88(2.3) | 10.9(2.5) | 18(3) |
| EDTA | 588(59.6) | 646(241) | 104(23.9) | 490(123) | 396(66) | |
| K-CX | 12.6(1.3) | < 0.010(< 0.01) | < 0.010(< 0.01) | 4.1(1.0) | < 0.010(< 0.01) | |
| P-L | 12.2(1.3) | < 0.010(< 0.01) | < 0.010(< 0.01) | 4.12(1.0) | < 0.010(< 0.01) | |
| 7日間 | 10%ギ酸 | 24(2.4) | 5.64(2.1) | 6.7(1.5) | 8.86(2.0) | 5.94(1) |
| EDTA | 352(35.7) | 364(129) | 630(145) | 430(98.6) | 318(53) | |
| K-CX | 12.4(1.3) | < 0.010(< 0.01) | < 0.010(< 0.01) | 4.22(1.0) | < 0.010(< 0.01) | |
| P-L | 12.8(1.3) | < 0.010(< 0.01) | < 0.010(< 0.01) | 4.04(1.0) | < 0.010(< 0.01) |
肺腺癌におけるDNA収量を示す。濃度はng/μLである。
( )内は処理なし(コントロール)のサンプルに対する回収率を示す(%)。
*10%ギ酸:10%ギ酸ホルマリン,P-L:プランクリュクロ
| C1 | C2 | C3 | C4 | C5 | ||
|---|---|---|---|---|---|---|
| 1日間 | 10%ギ酸 | 9.7 | 6.7 | 25.2 | 75.7 | 11.1 |
| EDTA | 30.6 | 65.9 | 102 | 140 | 218 | |
| K-CX | 0.7 | 0.6 | < 0.1 | < 0.1 | < 0.1 | |
| P-L | 0.7 | 0.6 | < 0.1 | < 0.1 | < 0.1 | |
| 3日間 | 10%ギ酸 | 2.9 | 2.5 | 3.2 | 4.4 | 2.5 |
| EDTA | 73.0 | 140 | 162 | 137 | 118 | |
| K-CX | 0.7 | 0.6 | < 0.1 | < 0.1 | < 0.1 | |
| P-L | 0.7 | 0.6 | < 0.1 | < 0.1 | < 0.1 | |
| 7日間 | 10%ギ酸 | 2.1 | 2.1 | 1.8 | < 0.1 | 3.2 |
| EDTA | 115 | 172 | 128 | 159 | 163 | |
| K-CX | 0.7 | 0.6 | < 0.1 | < 0.1 | < 0.1 | |
| P-L | 0.7 | 0.6 | < 0.1 | < 0.1 | < 0.1 |
大腸癌における処理なし(コントロール)と各種処理でのDNA収量の回収率を示す。
*10%ギ酸:10%ギ酸ホルマリン,P-L:プランクリュクロ
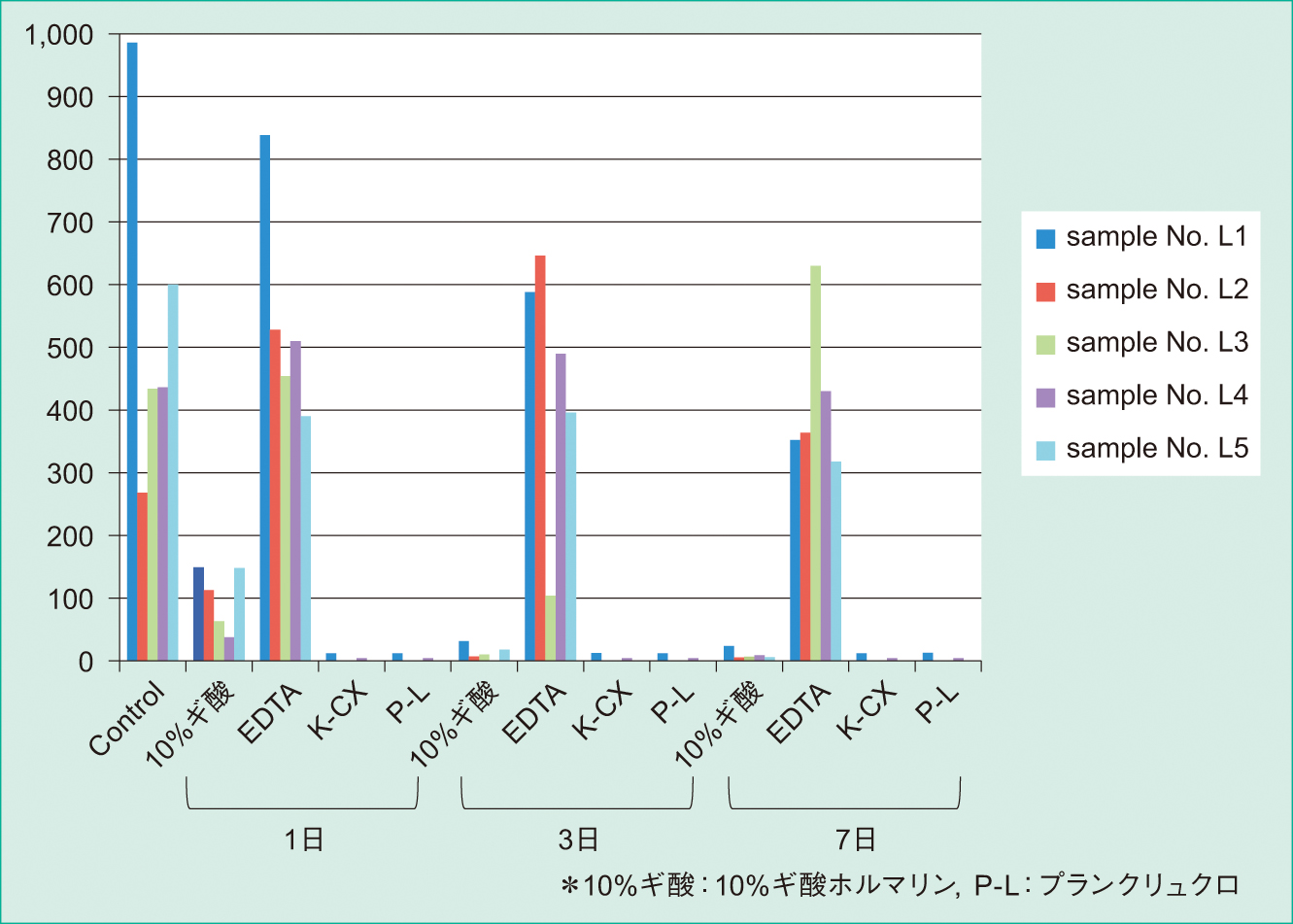
同じ症例でもサンプル間の差のバラツキは認められるもののEDTAでは,浸漬日数に関わらず処理なし(Control)と同様に十分な収量が得られている。10%ギ酸ホルマリンでは,1日の浸漬で若干の収量が得られているものの日数の経過とともに徐々に収量濃度が低くなっていた。K-CXとプランク・リュクロ脱灰液の強酸系の脱灰液では1日の浸漬だけでもかなりDNAの収量が低いことを示している。
K-CXとプランク・リュクロ脱灰液の強酸系脱灰液では,1日処理においてもDNA収量は極めて少なかった。10%ギ酸ホルマリンでは,強酸系脱灰液に比べDNA収量が多かったが,脱灰時間が長くなるにしたがってDNA収量は著しく減少した。一方,EDTAではすべての症例において,DNA収量が多く,脱灰時間に関しても7日間処理でもDNA収量の減少は緩徐であった。
3. PCRの増幅肺腺癌,大腸癌におけるPCR反応後のアガロースゲルによる泳動結果をFigure 4に示す。K-CXとプランク・リュクロ脱灰液では,両脱灰液ともに1日目の処理からバンドが認められずPCR増幅が確認できなかった。10%ギ酸ホルマリンではスメアー状の泳動結果となった。EDTAでは,処理日数に関わらず,100~400 bpのいずれも明瞭なバンドが確認できた。肺腺癌,大腸癌すべての症例において同様の結果を示した。

EDTAでは,処理日数に関わらず100~400 bpのいずれも明瞭なバンドが確認できる。10%ギ酸ホルマリンではスメアー状の泳動結果となった。K-CXとプランク・リュクロ脱灰液では,両脱灰液ともに1日目の処理からバンドが認められない。
コバス® EGFR 変異検出キットでEGFR変異の検索を行った結果をTable 5に示す。変異の有無に関わらず,EDTA処理した標本のみEGFR検索が可能であった。
| Sample No. L1 | 固定のみ | 1日 | 3日 | 7日 |
|---|---|---|---|---|
| 脱灰処理なし | 変異(−) | |||
| 10%ギ酸ホルマリン | Invalid | Invalid | Invalid | |
| EDTA | 変異(−) | 変異(−) | 変異(−) | |
| K-CX | Invalid | Invalid | Invalid | |
| プランクリュクロ | Invalid | Invalid | Invalid | |
| Sample No. L3 | 固定のみ | 1日 | 3日 | 7日 |
| 脱灰処理なし | L858R | |||
| 10%ギ酸ホルマリン | Invalid | Invalid | Invalid | |
| EDTA | L858R | L858R | L858R | |
| K-CX | Invalid | Invalid | Invalid | |
| プランクリュクロ | Invalid | Invalid | Invalid |
コバス® EGFR変異検出キットでEGFR変異の検索を行ったが,変異の有無に関わらず,EDTA処理でのみ検出可能であった。
HE染色や免疫組織化学染色における脱灰液の影響4)~6)については,比較的よく知られているが,DNAに対する脱灰液の影響についての報告は検索した限り少ない。Singhら7)はEDTA系やギ酸系の脱灰液を弱酸性系の脱灰液,塩酸や硝酸を使用している脱灰液を強酸系の脱灰液として分類し,それぞれの脱灰液に6~8時間浸漬した時のサンプルのDNA収量とCT値を比較している。弱酸性系脱灰液では,比較的DNA収量は多くCT値が低い。一方,強酸系の脱灰液ではDNA収量は著明に低く,CT値も高いと報告している。実際,われわれが行った検討結果でも,K-CXとプランク・リュクロ脱灰液の強酸系脱灰液では,DNA収量は極めて低く,PCR増幅においても一番小さいサイズの100 bpの増幅バンドも得られなかった。以上より,塩酸を使用している強酸系脱灰液は,DNAの断片化に大きく関与しており抽出や増幅を難しくしていると考えられた。したがってK-CXやプランク・リュクロ脱灰液といった強酸系脱灰液はFFPEにおける遺伝子検索に不適であることが示唆された。10%ギ酸ホルマリン処理においては,HE染色は比較的良好に保たれており,DNA収量も1日処理では強酸系脱灰液に比べ多かった。しかし,PCRでは10%ギ酸,K-CX,プランク・リュクロ脱灰液での増幅が得られない結果から,脱灰液による核酸の断片化による影響が示唆された。Brownら8)は,ギ酸とEDTA脱灰液における蛍光in situハイブリダイゼーション(fluorescence in situ hybridization; FISH)の検討を行っており,EDTAに関しては問題なく検出されているが,ギ酸に関しては,長時間の浸漬によりFISH法での検出が困難になるために,ギ酸の使用は最低でも24時間までの浸漬にとどめた方が良いとしている。また,今回行ったEGFR変異検出キットを用いた検索においては,10%ギ酸ホルマリンは,invalid(検出不能)となった。10%ギ酸ホルマリンは,脱灰効果も高く染色性も保たれていることから,実際の通常業務で多く使用されており取り扱いに関しても簡便であるため汎用されている。しかし,今回の我々の結果から,遺伝子検索には10%ギ酸ホルマリンの影響も考慮し使用を避けるべき傾向が示唆された。
日本病理学会のゲノム研究用病理組織検体取扱い規定(第3部 ホルマリン固定パラフィン包埋標本の適切な作製・保管方法,実証データ⑫)9)においてもプランク・リュクロ脱灰液と10%EDTAにおけるDNAの比較がなされており,プランク・リュクロ脱灰液においては,50 bpまで断片化されておりゲノム解析可能な品質のDNAの抽出は困難とされている。10%EDTAにおいては断片化が少ないゲノムDNAの抽出が可能とされており,今回,われわれが行った検討結果と一致した。また,同規定では,DNAの断片化について,ホルマリン固定時間が長期の場合においても300~500 bpの断片化を認めると報告されており,脱灰液と同様に留意が必要である。さらに,丸川ら10)のホルマリン加温固定によるDNAの断片化が報告されており,脱灰液のみならず過固定におけるDNAの断片化にも留意し,病理組織検体作製することが肝要である。
遺伝子変異解析に用いる組織標本において脱灰処理が必要な場合,EDTA を脱灰液として用いることが肝要である。
本研究計画は2018年1月18日東海大学医学部臨床研究審査委員会で承認された(承認番号:18R-239)。
本論文に関連し,開示すべきCOI 状態にある企業等はありません。