2023 Volume 63 Issue 2 Pages 79-85
2023 Volume 63 Issue 2 Pages 79-85
生物分子モーターは化学エネルギーを運動に直接変換する驚くべき分子機械であるが,その動作原理は本質的には理解できておらず,自在な応用のためにカスタマイズすることは困難であった.本稿では,ダイニンとDNA結合タンパク質を組み合わせ,DNAナノ構造に沿って動く新しいDNAモーターを構成した例を紹介する.

Biomolecular motors are remarkable molecular machines that directly convert chemical energy into movement. Yet the essential mechanisms remain unclear, making it difficult to customize them for versatile applications. In this paper, we present an example of a novel DNA motor that moves on DNA nanostructures by combining dynein and DNA-binding proteins.
近年,分子の検出や特性を調べるための分析技術の進歩はめざましく,日々新たな技術開発が行われている.一方で,分子の動的構造の設計や動作原理を理解した上で有用な分子を新しく生み出す技術は未だ発展途上である.もし,分子を自在に操ることができれば,分子レベルの機械を創ることによって,これまでマクロな機械では困難だった様々な仕事を行うことが可能になる.このような考えに基づき,1959年にファインマンによって分子マシンという概念が提唱された1).分子マシンとは,ナノメートルスケールの微小な空間において,一方向に回転したり移動したりなどの機械的な動きを通じて仕事を行う分子である.1959年当時は夢物語であった分子マシン構想は,巧みな有機化学合成技術によって具現化され,半世紀を経た2016年には,その将来性に対してノーベル化学賞が与えられた.このように,分子マシンを組み上げるための土台が徐々にできつつある.
分子マシンが仕事を行うには,ナノメートルスケールの微小な空間において機械的な動きを起こす必要がある.しかし,分子は熱運動によって互いに激しく衝突し合っており,言わば熱運動の嵐の中にいる.この嵐の中で,分子マシンには多数の分子がランダムな方向から毎秒一千万回ほど衝突するため,分子マシンが一方向に機械的な動作を行うことは容易ではないだろう.このような環境下において,分子がいかにして対称性を破り一方向性を獲得するかが重要である.
分子マシンの開発はこれまでに幾つかの分野で並行して行われてきた.ノーベル化学賞を受賞したFeringaらの成果の一つである「ナノカー」は,電圧パルスが印加されることによって,それ自体が一方向に移動する2).その原動力は,紫外線の照射によってオレフィン化合物とよばれる置換基の内部の二重結合が切断されて分子が回転できるような仕掛けである.彼らはナノカーのシャーシに巧妙に設計された車輪を組み込み,極低温・超高真空環境下で車輪を一方向に回転させ,ナノカーを動かすことに成功した.
また,DNAナノテクノロジーの分野では,2010年代初頭からDNAのみで構成された「DNA ウォーカー」の開発が盛んになった.DNAウォーカーが歩行するためのレール自体もDNAで構成されており,その表面にはDNAウォーカーの脚と結合できるような相補的な配列を持つ1本鎖DNAが多数生えている.DNAウォーカーの歩行は,DNAレールとの間の結合と解離の繰り返しによって起きる.酵素を原動力とした例では,まず,DNAウォーカーの脚がDNAレール上の相補鎖に結合し,2本鎖DNAを形成する.この2本鎖DNAを認識してDNAレール側の鎖のみを切断するような特殊な酵素を溶液中に加えておくと,酵素によってDNAが部分的に消化されたことでレールとの結合が不安定になり,その結果DNAウォーカー全体が熱運動で激しく揺れ動くことによって,DNAレール上の,まだ切断されていない相補鎖を探して結合し,一歩先に転移することができる.このように,ほとんどのDNAウォーカーは,レールDNA上の相補鎖を一つ一つ壊すことによって一方向にしか進めないように設計されている3).
これまでに様々な分子マシンの研究が進められてきたが,これらは自律性や運動の効率性といった点で未だ応用には遠い段階である.ナノカーは,光や電場などの刺激を外部から加える必要があるため非自律的であり,極低温・超高真空という特殊な環境下でないと作動しない.また,広義のDNAウォーカーの中には,その運動速度が80 nm s–1に達する高速なものがあるが,これは2次元平面に敷き詰められたDNAレールを破壊しながらランダムな方向に進むことと,レールが1回だけしか使用できないため応用が難しい4).これらの課題を解決するためには,すでに生体内で様々な仕事を行っている生物分子モーターの動作機構と設計方法に学ぶことが有効だろう.
生命活動には方向性のある「動き」が必須である.例えば,細胞内の特定の場所への物質の輸送,アクチン繊維を手繰り寄せて細胞自体を収縮させることや,細胞体自体を移動させるための鞭毛運動など,生命活動に欠かせない「動き」が数多く存在する.この「動き」の原動力となっているのが生物分子モーターである.生物分子モーターは,エネルギーを消費して細胞骨格(微小管やアクチン繊維)の上を一方向に運動し,多様な細胞内機能を果たしている.分子モーターは,その汎用性の高さから微小な駆動装置・モーターなどへの応用が期待されており,これまで数多くの研究がなされてきた.しかし,小さな生物分子モーターが熱運動の嵐の中でいかにして対称性を破り一方向に動いているのか,という点は未だ体系的に理解されていない.
近年の遺伝子操作技術や一分子計測技術などの発展により,たしかに生物分子モーターは,そのアミノ酸配列や構造,機能に至るまで様々なことが解明されてきた.しかし,いま偶然生き残っている分子モーターは,進化の歴史の中で成功した一部の種類の分子モーターと,そこから派生した子孫が大勢を占めている.そのため,「原理的に動作可能な分子モーターのバラエティー」という意味では,現存する天然の生物分子モーターはそのバラエティーのほんの一部でしかないと考えられる.しかも,これらのモーターは運動機能とは無関係な構造や運動機能以外の様々な機能も獲得している.そうすると,これらの生物分子モーターを分析した場合,どうしても個別のモーターに特有の構造や配列に注目せざるを得ないので,個々の構造に関する理解は進むが,どのように部品を構成すれば一方向性運動を起こすのか,という一般的な設計原理に関する情報には到達しにくい.この状況を打開するには,生物分子モーターとは無関係なタンパク質部品を組み合わせて人工分子モーターを作り,その中から機能するものとしないものを比較検討して,生物分子モーターの設計に必要な要素を帰納的に抽出するような構成論的手法が効果的である.
筆者らは,生物分子モーターが単純に前に進むという機能を実現するために3つの要素が必要であるという作業仮説を立てた.それは,(i)レールである細胞骨格との立体特異的な結合を行うこと,(ii)ATPの加水分解に伴う構造変化を利用して適切なタイムスケールで結合・解離を繰り返すこと,(iii)この結合・解離のプロセスのどこかにレールの一方の端へのバイアスを持つことである.これらを念頭に置いて,筆者らは新しい分子モーターを創ることを試みた.上記の(ii)のATPの加水分解に伴う構造変化については,天然由来の生物分子モーターの1種,ダイニンを用いた.ダイニンは,ミオシンやキネシンと異なり,微小管と結合する部分と,ATPを分解する部分とが明確に分けられるようなモジュール構造を持っているので,これらの要素それぞれを別の機能モジュールに置き換えることが可能であるという利点がある.
(i)の結合・解離を担うモジュールとして,筆者らはまずアクチン結合タンパク質を用いた.つまり,ダイニンがもともと持っている微小管と結合するためのモジュールの代わりに,アクチン繊維に結合するアクチン結合タンパク質由来のモジュールをダイニンと融合した5).その結果,この新規分子モーターは,本来は結合する相手ではないアクチン繊維と結合して,これを一方向に運動させる能力を発現することが分かった.ここまで思い切ったモジュールの組み換えを行っても運動機能を維持しているということは,今まで動作機構を考える上で前提とされてきたモジュール間の厳密な連携(例えば文献6)が,基本的な一方向性運動を生み出す上では必ずしも必要ではないことを示唆しており,これは非常に重要な結果である.
前述したように,生物分子モーターの設計原理を理解するためには,分子モーターを少しずつ無関係な部品へと置き換えていき,それらを詳細に分析することで,進化の文脈から切り離して考えていく必要がある.一方向性運動を創出する上で最も重要なのは,結合・解離のプロセスの中でレールの一方の端へのバイアスを持つという要素であると考えられる.しかし,アクチン結合タンパク質を融合した新規分子モーターに関して,これを詳細に調べようとすると,レールとモーターの間の結合面に大幅な変異を加える必要があり,この変異導入は容易にタンパク質を失活させてしまうため技術的に非常に困難である.また,微小管やアクチン繊維などの細胞骨格系タンパク質はGTPやATPを代謝する酵素であり7),8),レール側の構造変化が運動に影響を与えるという報告9)もあることからレール側の役割を無視できなかった.このような実験系の複雑さは,一方向性運動の創出メカニズムを理解する上で大きな障壁である.
この問題を解決するためには,レール側が構造変化せず,モーターとレールの間の非対称な結合と運動能との関係を系統的に調べることができる実験系を構築する必要がある.そこで筆者らは,DNA上を運動する新規分子モーターを構築することで,これらの課題を一挙に解決できないかと考えた.もともと遺伝情報を担うDNAは,安定な物質であり酵素活性を持たないので,分子モーターの運動に積極的には関与しないだろうと考えられる.このため,DNAレールは分子モーターの進化の文脈と切り離して考えることが可能である.また,DNAは1塩基レベルでの編集が可能なことや,近年のDNAナノテクノロジーの発展により,繊維状構造はもちろん複雑な3次元構造(DNAナノ構造体)を自在に設計可能である10).そこで筆者らは,DNAナノ構造体を新規分子モーターのレールとすることを着想した.
DNAをレールとして利用するため,筆者らはモーター側に天然由来のDNA結合タンパク質を融合することを考えた.細胞内には多くの種類のDNA結合タンパク質が存在するが,今回,分子モーターを作製する上で選定基準となったのは,DNAレールに対して,ある決まった向きに結合する性質である.2本鎖DNAは1本鎖DNA同士が逆平行に結合したものであり,この意味で極性を持たない繊維構造である.この逆平行の繊維に対して,回文配列を認識するようなDNA結合タンパク質は,DNAレールに対して互いに逆向きの2通りの方向を向いて結合することができる.そのため,本研究では非回文配列を認識するようなDNA結合タンパク質を用い,ヒト細胞質ダイニンの構造のうち,レールとの結合部位である微小管結合ドメインを,DNA結合タンパク質に置き換えた新規分子モーターを構築した(図1)11).

ヒト細胞質ダイニンの微小管結合部位をDNA結合タンパク質と取り換えて新規分子モーターを構築することを示した模式図.
DNAで構成されたレールとして,DNAタイルとよばれる技術を用いてDNAの二重らせん構造が束化した形のDNAナノチューブとよばれる中空の高次構造体を採用した10),12).このDNAナノチューブは,10本程度のDNA分子が互いに相補的な塩基配列の部分で結合し,DNAナノチューブの最小単位であるユニット構造を形成し,繊維状の構造体を形成する.このユニットには任意の配列を組み込むことが可能なので,DNA結合タンパク質の認識配列を組み込んでおけば,等間隔に新規分子モーターの結合部位を配置したレールを作ることができる(図2).このレールを用いて新規分子モーターの運動を顕微鏡観察した結果,DNAレールがスムーズに輸送される様子が観察され,しかもその速度は天然の生物分子モーターと比較しても遜色の無い220 nm s–1に達することが分かった(図3A, B).運動速度はATPの代謝速度とは明確な相関が無く,運動速度は例えばDNAからの解離など,他の過程の速度定数で決まっている可能性が考えられる(図3C, D).

12本のDNA2重らせん構造が束化したDNAナノチューブ(上)と10本のオリゴDNAによる基本最小単位であるユニット(設計図,下)を示した模式図.配列の近くに引いた線の色は異なるDNA鎖を示し,矢印の方向は5ʹ→3ʹの方向を表している.
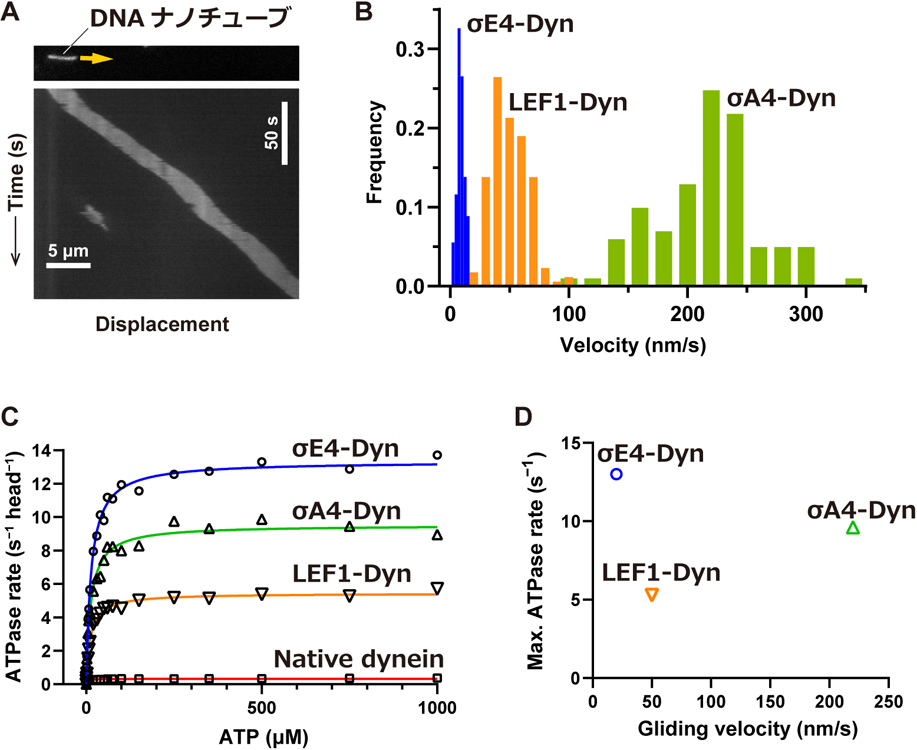
(A)ガラス面上に新規分子モーターを固定し,その上に結合した1本のDNAナノチューブが滑走する様子を示したキモグラフ.(B)3種類の新規分子モーターの速度分布ヒストグラム.(C)DNAナノチューブ.または微小管非存在下における新規分子モーターと野生型細胞質ダイニンの定常状態のATPase活性を示したグラフ.(D)運動速度と最大ATPase代謝速度の関係をプロットしたグラフ.
DNAレールの合理的な設計は,既存の細胞骨格ではできないことの一つである.そこで,運動特性に影響を与える可能性が高いと考えられるレールの剛性・結合部位の向きなどを制御したDNAナノチューブを幾つか設計した.まず,レールの剛性と運動特性の関係を調べるために,4本(4-helix),10本(10-helix),および20本(20-helix)の二重らせんが束化した3種類のDNAナノチューブを作製した.レールの硬さを示す指標である持続長は,10-helixのDNAナノチューブでは16.8 μmであることが報告されており13),報告されているアクチン繊維の持続性(17.7 μm)に近い14).また,4-helixのDNAナノチューブの持続長は2.0 μm以下であるとされている13).グライディングアッセイにおける機能評価では,4-helixのDNAナノチューブは,10-helixと20-helixに比べて柔軟性が高く,基板表面で大きく蛇行することや,伸縮する様子が観察されており,運動は滑らかではなかった.一方で,10-helixと20-helixのDNAナノチューブは,天然由来の分子モーターの運動と同様,滑らかにDNAナノチューブが滑走している様子が観察された(図4A, B).この実験により,モーターの運動にレールの剛性が大きな影響を与えることが分かった.

(A, B)10-helixと20-helixのDNAナノチューブの模式図.下部の画像は,DNAナノチューブの動きを時間ごとに色分けし,すべてを重ね合わせて時系列的に視覚化したもの.(C)様々なイオン強度での10-helixのDNAナノチューブの速度,および20-helixのDNAナノチューブの速度を示したグラフ.(D)極性DNAナノチューブに対して,認識配列のみを反転させた際の新規分子モーターの動きを示した模式図(左),新規分子モーターにより駆動される2種類の極性DNAナノチューブの連続画像(中央),新規分子モーターの運動方向と速度分布ヒストグラムを示したグラフ.
先行研究では,生物分子モーターが一方向性運動を実現する方法として主に2つの説が提案されている.一つはパワーストローク説で,これは分子モーターがATPの加水分解に伴って大きな構造変化を起こすことで熱運動に打ち勝って動作すると説明されることが多く,現在の生物の教科書では主流である.もう一つはブラウンラチェット説で,これは逆に分子モーターが熱運動を動力源として利用し,レールから解離しやすい方向に差を付けるなどの方法で熱運動の方向を制限する(整流する)ことで動作すると説明される.これらの2つの説は一見すると対立しているように見えるため二者択一的な議論が行われてきたが,一方で両者がお互いに排他的なのかどうかについても議論され15),16),最近では両者が両立できることも指摘されている17).しかし,現状は仮説を検証するための手段や方法論が限られているため,実際に生物分子モーターがこれらをどのように使い分けているのか,などの議論は行き詰まっている状況であると思われる.
分子モーターの運動方向は,結合部位に対するモーターの結合面の向きが決めていることは先行研究で示唆されてきた5),18).ただし,これは結合面そのものの向きを変えた実験ではなく,モーター側に変異を入れて,上記のパワーストロークの向きを変えるような実験によって示されていた.ブラウンラチェットモデルにおいて運動方向を決めるのは構造変化の向きではなく結合面の向きであり,これらは注意深く分けて考える必要がある.これまでは,モーター側の構造の向きを反転させることはできても,レール側の結合部位の向きを反転させることはできていなかった.というのも,結合部位を反転させるには遺伝子工学的手法を用いて細胞骨格系タンパク質の中のモーター結合部位を反転させるような大掛かりな変異を導入する必要があり,このような変異を細胞骨格に導入することは現状では困難だからである.一方,DNAで構成されたDNAナノチューブでは,認識配列を逆向きに配置するだけで容易にモーターとの結合面のみを反転させることが可能である.そこで筆者らはDNAナノチューブを構成するすべての配列のうち,認識配列のみを反転させて新規分子モーターの運動方向を観察した.このとき,DNAナノチューブ内での認識配列の向きを明確にするために,DNAナノチューブの端を,レールとは別の蛍光色素で可視化し,極性マーカー付きのDNAナノチューブを作製した19).その結果,認識配列の向きだけを反転させると,その運動方向が完全に反転することが確認された(図4D右).これにより,分子モーターの運動方向は,モーター結合部位の結合面自体の向きに依存していることが確認された.もちろんこの結果は,結合面が反転した結果,パワーストロークの向きが反転し,その結果として運動方向が反転したことを示しているとも考えられるが,一方で筆者らの未発表のデータでは,モーター側の改変によって,パワーストロークの向きを変えずに結合面だけを反転させることによっても運動方向を反転させることが可能であることを示す実験結果も得られている.このように,改変ダイニンモーターの実験系の大きな利点は,パワーストロークの向きと結合面の向きを独立に変えることができる点である(パワーストロークの向きを変える実験は文献18を参照).今後,改変ダイニンモーターを使った構成的実験によってパワーストロークとブラウンラチェットのそれぞれが,どの程度,どのように一方向性運動に貢献しているかなど,これまで不明瞭だった運動メカニズムの詳細が明らかになると期待される.
これまでに,性質の異なる幾つかの種類のDNAナノチューブを作製し,レール側の配列や構造を変えることで新規分子モーターの運動制御が可能であることを実証した.ここで少し分子モーターの工学的な応用に触れる.第4節で述べた細胞骨格の不均質性や不安定性の問題は,運動メカニズムを解明するための研究だけでなく,分子モーターを工学的に応用する研究を進める場合にも生じる.細胞骨格のダイナミックな性質は,細胞内輸送や染色体分離といった多様な細胞内プロセスに不可欠な性質である.しかし,これらの性質は,正確なレールの制御や再現性が必要とされる工学的な応用にとっては不都合である.一方,DNAは化学的に安定な物質であり,かつ,これまでの実験から分かるように精密な構造設計や1塩基単位の改変が可能である.そこで,筆者らが開発した新規分子モーターとDNAレールのシステムを利用すれば,細胞内で実際に行われているような分子の自動的な選別や統合といった仕事を,単純化した形で再構成できるのではないかと考えた.
分子の選別や統合を行うにはレールの分岐が必要となる.そこで,3本のDNAナノチューブが1点でつながった分岐構造を検討した.先行研究で,すでにDNAナノ構造体の1種であるDNAオリガミ技術を使ってY字型のアダプターを作製して重合核とし,ここからDNAナノチューブを重合することで枝分かれを作るようなY字型の構造体が報告されており20),筆者らはこの構造体を改変して利用した.具体的には,このY字型のレールの中心から伸びる異なる3本のDNAナノチューブに,異なる2種類の新規分子モーターの認識配列を埋め込み,各レール上で走るモーターの種類や運動方向を制御した.このレール上で2種類のモーターを同時に動かすことで,分子の選別や統合を行う「統合器(integrator)」や「選別器(sorter)」を設計・作製した(図5A, B).2種類のモーターをそれぞれA,Bとすると,まず1本目のDNAナノチューブ(A-レール,図5Aの画像では緑色)にはAに対応する認識配列と蛍光色素が組み込まれている.2本目のDNAナノチューブ(B-レール,図5Aの画像ではピンク色)にはBに対応する認識配列と蛍光色素が組み込まれており,3本目のDNAナノチューブ(AB-レール,図5Aの画像では白)にはAとBの両方の認識配列と,両方の蛍光色素が組み込まれている.選別器と統合器の違いは各モーターの運動の向きだけである.つまり,3本のDNAナノチューブがつながっている合流点に対して,それぞれのDNAナノチューブに組み込まれる認識配列の向きを適切に変えるだけで,各モーターの運動方向が制御され,選別器や統合器を作製できる.

(A, B)選別器と統合器を構成するY字型のレールを蛍光顕微鏡により観察した画像と,2種類の輸送複合体がY字型のレール上で一つのレールへと荷物を集めて統合する,または2つに分けて選別する様子を時系列で示した画像.(C, D)合流点や分岐点に到達した輸送複合体のその後の振る舞いを,停止,解離,合流に分類し,その割合を示したグラフ.
実際に,2種類の新規分子モーターが分子の選別や統合を行うことができるかどうかを顕微鏡下で調べた.荷物として使用したのは長さ400 nm程度の棒状の構造を持つ2種類のDNAオリガミで,これらのDNAオリガミは区別できるようにATTO 550とATTO 647Nの2種類の蛍光色素で標識した.それぞれの荷物には,2種類の新規分子モーターであるAとBをそれぞれ結合させた.この2種類の輸送複合体AとBは,それぞれに対応した認識配列が組み込まれたDNAナノチューブにのみに結合して運ばれることが確認され,これらのモーターの間には,荷物同士の非特異的な結合以外にはクロストークは観察されなかったことから,選別器や統合器として機能することが確認された.
既知のモジュールを組み合わせる構成的な方法による研究は,系統的にレール側の配列を改変して,多種類のモーター-レール間の組み合わせから数多くの機能-構造相関データを取得できるため,これまでの分析的な研究とは異なる角度からタンパク質モーターの作動原理を探るための強力な手法になると期待される.ただし,上記のダイニンの結果は重要な示唆ではあるが,ダイニンのモーター部位は複雑で大きいため,未だ多くの部分がブラックボックスである.今後,一方向性運動に必要な要素を検証するためには,ATPの加水分解部分を非モーター分子から構成することや,ATP加水分解以外の酵素反応を一方向性運動に変換することにチャレンジすべきだと考える.
工学的な見地からは,複数のモーターによる分子選別・統合を自律的に行えることを示したことは大きなブレイクスルーと言える.本研究で作製された新規分子モーターは,DNAレール上で繰り返し運用できる最速の人工分子モーターであり,これまで動作に分単位の時間が掛かっていた静的なDNA構造体に,秒以下の動的な構造変化などの性質を与えることを可能とした.したがって,本研究は,分子モーターの動作・設計原理への理解を深めるだけでなく,生体分子の輸送システム,形状を環境に応じて変化させることができるスマート材料,分子を用いた計算,テーラーメイドなポリマーを合成するナノ工場など,分子マシンを用いた新たな産業への道を開く可能性があると考えている.