2022 Volume 62 Issue 5 Pages 303-304
2022 Volume 62 Issue 5 Pages 303-304
細菌のリボソームは,2つの保存されたタンパク質RRFとEF-Gの協働によりサブユニットにリサイクルされるが,分子基盤は未だ不明である.本稿では,EF-G,RRF,2つのtRNAが結合した70Sリボソーム複合体のX線結晶構造をもとに,リサイクルにおけるtRNAとRRFの積極的な役割を紹介する.

リボソームにおけるタンパク質合成,すなわち翻訳は,開始・伸長・終結の三段階で構成される.翻訳終結はmRNAの終止コドンがリボソームのAサイトに到達し,終結因子がポリペプチド鎖の放出を促進することで起きる.このときリボソームにはPサイトとEサイトにmRNAと脱アシル化tRNAが残される.バクテリアでは,翻訳伸長因子EF-Gとリボソームリサイクリング因子RRFの協働によりリサイクルされる.リボソームを再利用して新たな翻訳を開始することはタンパク質合成の必須ステップで,GTP加水分解エネルギーを用いて70Sを30Sと50Sサブユニットに分離する.Pサイトに脱アシル化tRNAが存在すると,サブユニットへの分離が15倍以上速く進み1),効率的なリサイクルにはtRNAが必要であると考えられている.しかし,tRNA非結合型2),Eサイト結合型3),p/Eハイブリッド結合型4)のリボソーム・RRF複合体の結晶構造からはサブユニットへの分離におけるtRNAの役割は説明しがたい.
近年の翻訳伸長複合体の構造研究から,EF-Gは転位前のリボソームと結合するためにコンパクトな構造を取ることが明らかになった5),6).その後,GTP加水分解によってEF-Gが拡張した構造に変化すると,リボソームの構造変化を伴い,mRNAやtRNAの転位が起こる.一方,RRFやEF-Gが結合したリボソームをサブユニット分離前の状態で捕捉して構造決定することは依然として困難である.そこで我々は,RRFとEF-Gによるリボソームリサイクリングの際に,EF-Gが一時的にコンパクトな構造を取り,転位前のリボソームに結合するのではないかと考え,目的複合体のX線結晶構造解析を試みた.本稿ではRRF,GDP結合型EF-G5),脱アシル化tRNAおよびAサイトに終止コドンを含むT. thermophilus (Tth) 70Sリボソーム複合体の結晶構造を紹介する.
先行研究を参考に,リボソームリサイクリング複合体はリボソームタンパク質bL9のN末端ドメインとTthEF-GのN末端との融合体を用いて再構成した2).この戦略は結晶中において融合体のbL9領域が,内在性bL9欠損70Sに結合し,GDP結合型・抗生物質非結合型EF-Gを隣接するbL9欠損70SのGTPase結合領域に捕捉するものである5),7).
今回の結晶構造にはリサイクルに必要なEF-G,RRF,mRNA,tRNAを含むすべての構成要素が確認された(図1a).RRFの全体構造は先行研究の結晶構造中のRRFの全体構造と類似している2)-4).EF-Gは先行研究同様にコンパクトな構造を取っており5),ドメインIIIとVが,RRFのドメインIIと形状相補的に相互作用している(図1b).この結果はEF-GのドメインIIIとVがRRFのドメインIIと相互作用する過去のCryo-EM再構成の結果と一致する8).

EF-Gが存在しないRRF・リボソーム複合体の構造と比較すると,RRFのドメインIは同じ位置にあるが,EF-Gが結合することでRRFのドメインIIが23S rRNAのヘリックスH69に向かって約28°回転し,リボソームタンパク質uS12とH69の間に挟まる.これに伴い,H69の先端が,H69とサブユニット間架橋を形成する16S rRNAのヘリックスh44に向かって約8 Å移動することで,RRFのドメインIIが配置される隙間が形成される(図1c).
このリボソーム複合体で最も注目すべき点は,Pサイトの脱アシル化tRNAの結合状態である.RRFとの立体的な衝突を避けるために,P-tRNAPheはアンチコドンステムループ(ASL)のらせん軸に沿って約30°曲がり,アクセプターステムが約22 Å移動して,tRNAのCCA末端が50SのPサイトとEサイトの中間に位置する(図1d, e).これはRRFのドメインIの先端が23S rRNAのP loopと相互作用することで,CCA末端とP loop間に通常形成される塩基対が破壊され,CCA末端のリン酸骨格が23S rRNAのヘリックスH74とH80によって締め付けられて,それ以上の動きが阻害される(図2b).CCA末端のA76が180°反転していることから,CCA末端と23S rRNAの間に好ましくない相互作用が生じていることが示唆される(図2c).以上のようにtRNA上部で構造変化するにも拘わらず,アンチコドンは基本的に30SのPサイトに留まる.我々の知る限り,このようなtRNAの結合状態は未だ報告されておらず,リボソームリサイクリングにおける機能的意義を考慮して,p/R-tRNAと呼称する(図1d, e).
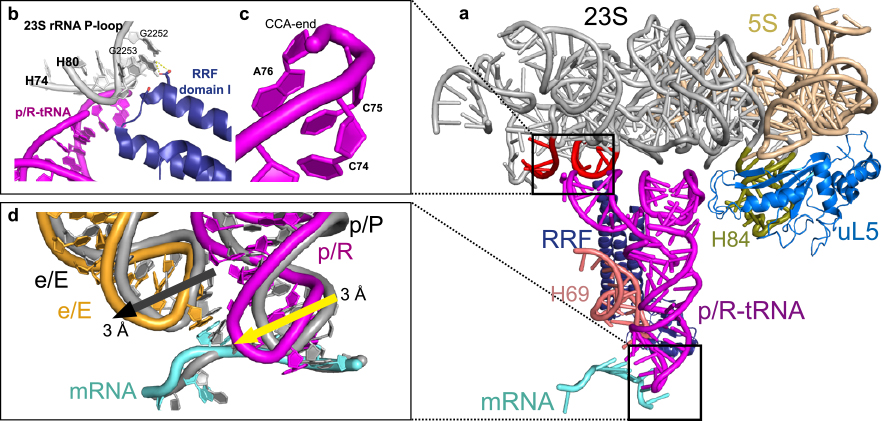
a,p/R-tRNAとその周辺のリボソーム構成要素.b,RRFのドメインIと23S rRNAのP-loop間相互作用および23S rRNAのH74とH80間によるp/R-tRNAのCCA末端への締め付け.c,その締め付けの際に起きるCCA末端のA76の反転.d,古典的な状態で結合したtRNAと比較したリサイクリング複合体のPおよびEサイトにおけるASLの動き(文献7より改変).
RRFを介したp/R-tRNAの構造変化は,30S側のPサイトのASLに伝わり,コドン・アンチコドン塩基対を弱める7).脱アシル化tRNAがPサイトからEサイトへの転位中に起きるp/Eハイブリッド状態とは対照的で,p/R-tRNAのASLはEサイト側に3 Å程度シフトすることで,コドン・アンチコドン塩基対はほぼ崩壊し,非結合原子間の平均距離は4 Åである.同様にE-tRNAのASLも出口側に3 Åほど移動していることが観察された(図2d).
今回の構造解析から,EF-G,RRF,脱アシル化tRNAが協働するリボソームリサイクル機構が明らかになった.翻訳終結後のリボソームにコンパクト構造のEF-Gが結合すると,RRFがサブユニット間架橋の隙間を認識する構造になる(図1c).P-tRNAはRRFとの構造的衝突を避けるためにp/R結合状態へ変化し,50S間の一時的で不安定な相互作用によりp/R-tRNA CCA末端が圧縮されると,50Sとの間に張力が生じ,サブユニットが分離しやすくなるバネのような機能が示唆される(図2b, c).この構造変化が30S側のASLに伝播し,コドン・アンチコドン塩基対を弱めることでリボソームからの解離が促進される.このモデルは,脱アシル化tRNAの存在下でサブユニットの分離率が上昇するという報告に根拠を与えるものであると示唆される.
本研究は,著者がUniversity of Texas Medical Branch, Galveston, TXにおいて博士研究員としてMatthieu G. Gagnon, Ph.D., PI(Assistant professor)に師事した期間に,上海・復旦大学のJinzhong Lin, Ph.D., PIおよびDejian Zhou, Ph.D.と共同で行ったものである.この場を借りて厚く御礼申し上げます.