2022 年 62 巻 6 号 p. 348-350
2022 年 62 巻 6 号 p. 348-350
我々の開発した光単離化学(Photo-Isolation Chemistry: PIC)という技術は,関心領域(ROI)の高深度トランスクリプトーム情報を高解像度の光照射によって抽出できる.大小さまざまなROIに対応できるため,マウス脳の領野,マウス胚の微小細胞集団や,細胞内の非膜型オルガネラの高深度トランスクリプトーム解析に活用できる.

多細胞生物は多様な組織や細胞タイプから構成され,それらは空間的に整然と配置される.その細胞タイプの性質は遺伝子発現によって規定されるため,多細胞システムの理解には位置情報と遺伝子発現情報を紐づけたトランスクリプトーム解析が必要である.従来,in situ hybridizationや免疫染色によって組織内における個々の遺伝子の空間的な発現パターンが解析されてきたが,空間的な網羅性をさらに高めた解析技術の開発がこの数年で急速に加熱している.それらは空間トランスクリプトーム技術と総称され,おもに空間網羅タイプと局所深読みタイプの二つに大別される.空間網羅タイプの技術では組織切片の遺伝子発現を広域にわたって空間網羅的に解析する.MERFISHやseqFISH+に代表されるこの技術では,連続的にin situ hybridizationを繰り返すことにより,細胞内にある多種類のRNAの分布を一分子レベルで可視化できる1),2).また10X Genomics社から商用化されているVisiumに代表される技術では,空間バーコード配列を含むオリゴDNA基盤に組織切片を貼り付ける.これによって細胞内のmRNAが基盤上のオリゴDNAとアニールして逆転写反応を進める.そのcDNAと空間バーコード配列をシーケンスすることで,遺伝子と空間情報を対応づけることができる3).いっぽうであらかじめ関心領域(ROI)が決まっている場合は,その遺伝子発現プロファイルを高深度に解析する局所深読みタイプの技術が有用である.これは空間的な網羅性に劣るが,局所領域の高深度トランスクリプトーム解析に適している.レーザーキャプチャーマイクロダイセクション法では,高出力レーザーでROIを物理的に切り出してトランスクリプトーム解析をおこなう.より高精度にROIを定義するために,近年では光照射によって化学的な変化を与える手法が開発されている.なかでも我々が開発した光単離化学(Photo-Isolation Chemistry: PIC)は,サブミクロンレベルの分解能で光照射したROIから高深度のトランスクリプトーム情報を抽出できる4).本稿ではPICの原理とスペックについて概説し,実施のためのコツについても紹介する.そのほかの空間トランスクリプトーム技術の詳細については我々の他誌への寄稿5)をご覧いただきたい.
PIC によるRNA-seq技術はROIのトランスクリプトーム情報を光照射によって引き出すことができる.その原理は以下の4ステップからなる(図1).①光ケージ化合物(NPOM)を付したオリゴDNAを組織切片に滴下し,すべての細胞内で逆転写反応する.②ROIのマーカー分子に対する抗体で免疫染色する.③ROIに限定して光照射することで,cDNAにつながるオリゴDNAからNPOMが脱離する.④切片のすべての細胞のライセートを回収してライブラリ合成する.まず,cDNAに連結された一本鎖オリゴDNA をfill-in反応する.光照射されたROIに由来するオリゴDNAからはNPOMが脱離しているため,fill-in反応によって末端のT7プロモーター(図1のオレンジの配列)までが二本鎖DNAとなる.そのcDNAだけがT7 RNA polymeraseによるin vitro transcription法で増幅され,シーケンスできる.

PIC RNA-seq法の原理.
脱ケージのための光は対物レンズを通してROIに射出される(詳細は後述)ため,対物レンズの倍率を変えることで大小さまざまなROIに対応できる(図2).たとえば,マウス脳の海馬のCA1,CA3,歯状回(DG)領域,E14.5マウス胚の神経管における背側または腹側ドメイン,E10.5マウス胚における生殖巣に移動中の始原生殖細胞などに光照射して,その高深度トランスクリプトーム解析に成功している.さらに小さなROIとして,我々は核スペックルという1 μm以下の核内構造のトランスクリプトーム解析に挑戦した.これはRNAとタンパク質が液-液相分離によって凝集した非膜型オルガネラであるが,サイズが小さいうえに構造が不安定であるため,物理的・生化学的に単離することは難しく,そのトランスクリプトーム解析は前例がなかった.我々はNPOMを付したオリゴDNAによりHeLa細胞で逆転写反応後,核スペックルマーカー(SC35)に対する抗体で免疫染色し,核スペックルだけに光照射することで,そこに特異的に局在するRNA分子を検出した.その中には核スペックルを構成するMALAT1転写産物のほかにも1,000種類ほどのRNAが検出されたため,PIC法における空間分解能と検出深度の高パフォーマンス性が示された.
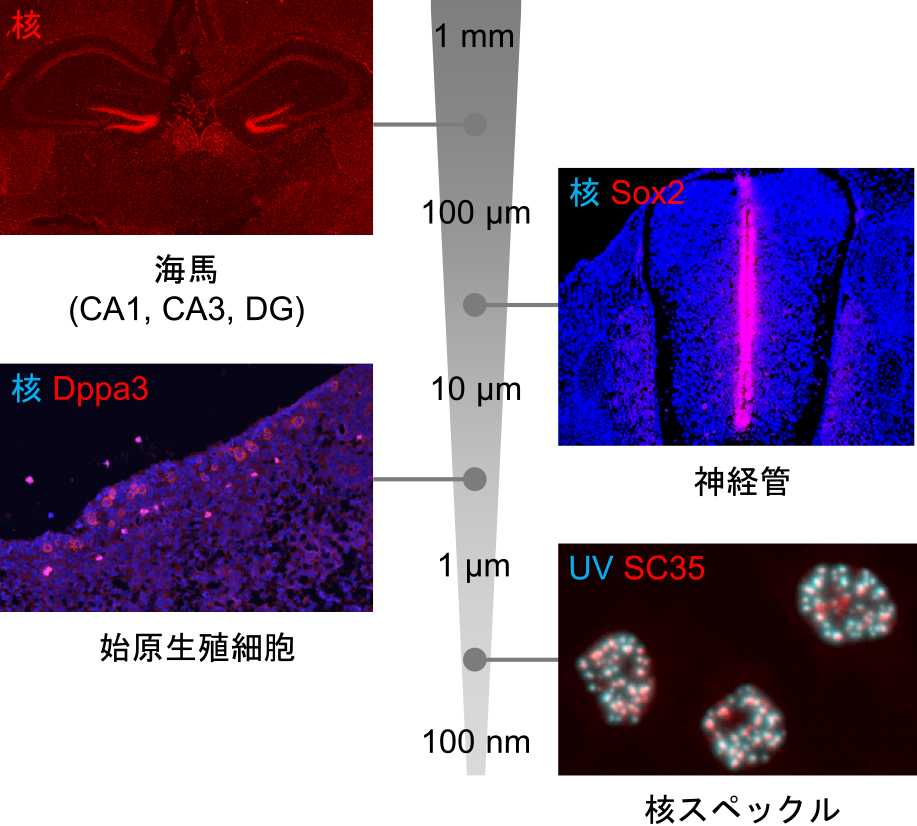
PIC RNA-seq法の実施例.
当初,PIC RNA-seq法は新鮮凍結切片に対して開発されたが,最近我々は臨床サンプルに多用されるホルマリン固定した凍結またはパラフィン切片にも適用できることを示した6).その肝となるのが組織切片に対する前処理で,0.1 N HClに5分間浸す,または70度のTris-EDTA緩衝液(pH 8.0)で1時間加熱する(図3).これによりmRNAをとりまくタンパク質などを緩め,細胞内逆転写反応が促進すると考えられる.未固定組織はHCl,TEともに同等の効果が得られるが,ホルマリン固定組織はTEのみ有効である.新鮮凍結切片は検出感度に優れるが,組織形態が崩れやすいうえに,抗原の保持性が低いという難点がある.これはROIを可視化するための免疫染色において注意すべき点で,ほかの分子と相互作用しにくい抗原(好例はGFP)は未固定切片から流失してしまい検出されなくなる.その場合は別のROIマーカー分子で染色するか,ホルマリン固定した組織を使うとよい.したがって,初めての組織では未固定組織と固定組織の両方を準備して透過処理し,免疫染色とライブラリー合成のテスト実験をおこなう.組織形態,免疫染色像,ライブラリ収量のバランスを考慮し,本番用のサンプル条件を決定する(図3).
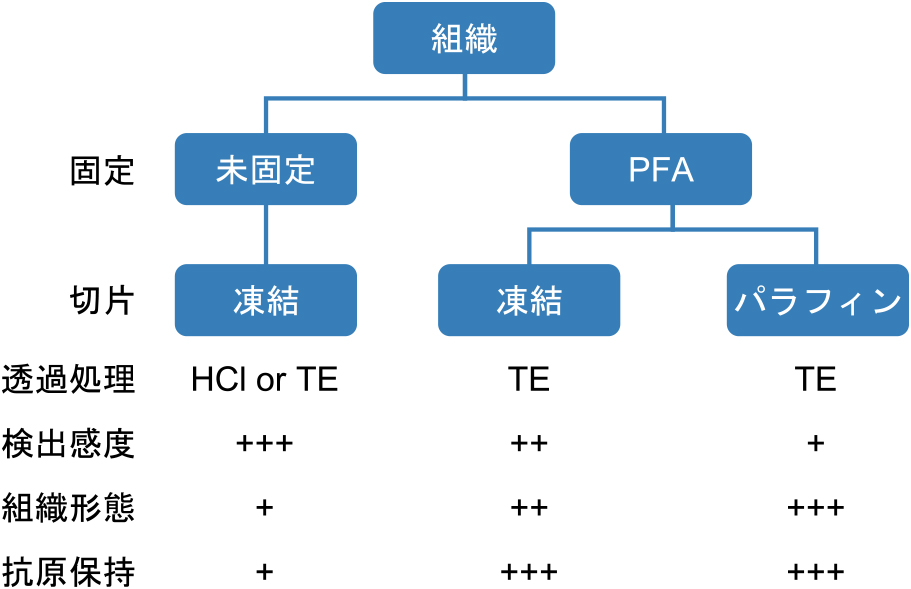
サンプル条件の選択チャート.
PICで用いられる,NPOMでケージした逆転写プライマーは外注によって容易に合成できる.詳しくは我々のプロトコール論文6)に記載しているが,NPOMでケージされたチミンを購入し,それをオリゴハウスに送付して合成してもらう.
NPOMは365 nmの光照射によって脱離する.通常の蛍光顕微鏡であればDAPIやHoechstを励起するためのフィルターセットで落射光をROIに当てればよいが,光の形状は円形に限られる.またレーザー顕微鏡では任意の形状の光を照射できるが,365 nmに対応した顕微鏡は入手が困難である.そこで我々はdigital mirror devise(DMD)を使用している.これは数百万枚の微小ミラーの集合体であり,それぞれが印加された電圧に応じて角度を変える.パソコンで描いた図を電圧情報としてDMDにロードすればその通りのパターンの光が反射されるため,任意の形状のROIに光照射できる.実際の実験ではまず,蛍光顕微鏡に内蔵された光路系で免疫染色像を撮影する.このとき,420 nm以上の波長域で励起するよう注意する.次にDMDに付属したソフトでROIを描き,365 nm LED光源をオンにすると目的の領域が脱ケージされる.その後,組織ライセートをチューブに回収してライブラリ合成するが,一般的な分子生物学実験手法にしたがって容易におこなえる.
本稿で紹介したように,PICは局所領域のトランスクリプトーム情報を高解像度かつ高深度に検出することができる.しかし,局所領域の遺伝子発現がどのように制御されているか?という問題が次の課題となる.現在,我々はオープンクロマチン領域や,タンパク質-ゲノム相互作用を読み取るための技術にPICの転用を試みている.これにより,局所領域の遺伝子発現を司るエンハンサー配列や転写因子結合などをゲノムワイドにプロファイリングし,空間的な遺伝子制御の解明に活用できると期待している.