遺伝子網羅的な計測によりデータ駆動的に規定された個々の細胞の状態を,AIを用いて細胞画像から推定した.ロボットの開発による自動化によりデータ取得を効率化し,この推定を実現した.

多細胞生物の成り立ちを理解するために,一つ一つの細胞の特徴を解析することは重要である.そのアプローチの一つとして,個々の細胞内のRNAの種類と数を計数できる1細胞RNA sequencing(RNA-seq)が近年発展してきた1).RNA-seqでは,遺伝子網羅的な解析(トランスクリプトーム解析)からデータ駆動的に特徴を見出し,細胞の種類や状態を規定することができる.しかしながら,1細胞RNA-seqでは基本的に計測対象の細胞を破砕する必要があるので,経時的な計測は難しい.
一方で,1細胞の経時的な計測ができる手法として顕微鏡イメージングが挙げられる.生きたままの細胞を継続的に観察できるため,細胞の動的な特徴を捉えることができる.しかし,RNA-seqのように遺伝子網羅的なデータの取得はできない.例えば,蛍光タンパク質を用いて遺伝子発現を観察する場合,波長により区別できる蛍光タンパク質の種類に制限があるからである.
将来的に,分子網羅的かつ経時的な計測の実現が期待されるが,ハードルは高い.一方で,AIを用いたアプローチが可能性を拓くという期待がある.近年発展しているAIは,生物学研究での応用も進んでいる.細胞や組織を対象にした研究では,深層学習を用いて細胞の輪郭を効率よく同定したり2),組織の病変部位を推定することなどが行われている3).これらには教師あり学習という手法が使われることが多い.教師あり学習の単純な例としては,がん細胞と正常細胞の2種類を用意し,画像とラベル(“がん細胞”や“正常細胞”)の組み合わせをAIで学習し,その後に画像だけをインプットしてラベルを推定する,というものが挙げられる.
本稿では,分子網羅的かつ経時的な計測の第一歩として,網羅的計測で規定された遺伝子発現状態を教師あり学習を用いて細胞画像から非侵襲的に推定した成果4)を紹介し,その発展性や展望を述べる.
細胞画像から網羅的計測により規定される細胞状態の推定に向けて,細胞画像に紐づいた遺伝子網羅的な計測結果を得るために,光学顕微鏡で観察した細胞のRNA-seqを実施した.学習には多くの細胞のデータが必要と予想されたため,まず,顕微鏡で観察した細胞を自動で分取する装置,ALPS(Automated Live imaging and cell Picking System)を開発した(図1).ALPSは顕微鏡と細胞分取装置を統合したシステムで,細胞の位置の同定,細胞画像の取得,細胞の特徴による選択,細胞分取,細胞のチューブなどへの吐き出し,を自動で繰り返し行うことができる.ALPSを用いて,3種類の培養細胞(白血病細胞,T細胞,造血前駆細胞)を混ぜたサンプルをタイムラプス観察した後に各細胞を分取し,96ウェルプレートの個々のウェルに1細胞ずつ吐き出し,その後,1細胞RNA-seqを実施した.RNA-seqに必要なライブラリ準備の工程も自動化してハイスループット化し,このサンプルでは1,008個の細胞のデータセットを得た.
得られたデータセットについて,トランスクリプトームの結果に基づいてクラスター解析したところ,予想通りに三つのクラスターが識別された(図2).このデータ駆動的にクラスター化したグループ(ここでは細胞種に相当)をラベルとし,まず,データセットの約95%の細胞を用い,時系列細胞画像からラベルを推定する深層学習モデルを作成した.その後,残り約5%の時系列細胞画像について,得られたモデルを用いてラベルを推定させたところ,細胞画像から81%の正答率で3種類の細胞種(白血病細胞,T細胞,造血前駆細胞)を識別できることを確認した.
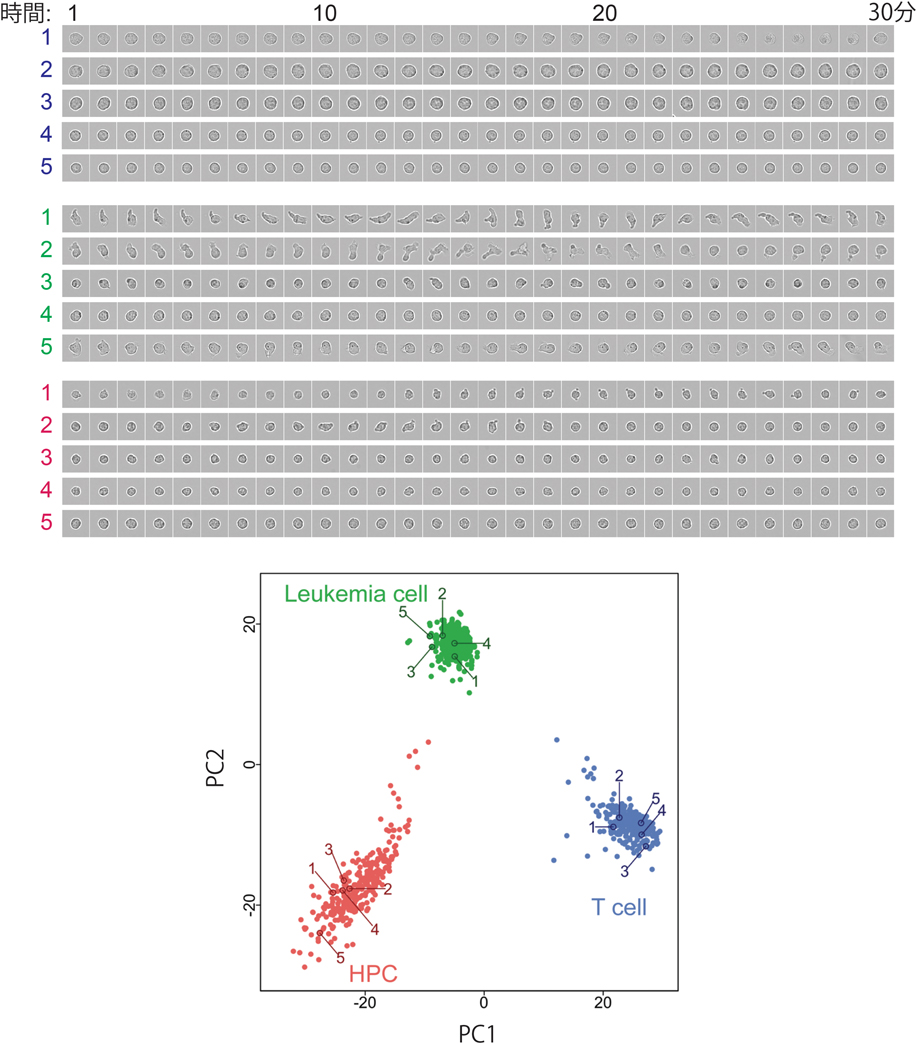
タイムラプス観察で得られた3種類の培養細胞(本文参照)の画像(上)と,RNA-seqにより得られた各細胞の網羅的遺伝子発現量を用いた主成分分析(下).主成分分析にて示したすべての細胞について画像データを取得しており,番号は例として同じ細胞からのデータセットであることを示している.PCはPrincipal Componentの略.一部は文献4からの改変.
さらに,プライマリ細胞において,本アプローチの有効性を示した.具体的には,マウス末梢血から細胞を調製し,先述の培養細胞の場合と同様に,ALPSを用いてタイムラプス観察した細胞を分取し,分取した各細胞のトランスクリプトームをRNA-seqにより計測した.その結果,時系列細胞画像から,データ駆動的に決定した3種類の免疫細胞種(B細胞,CD4+T細胞,CD8+T細胞)を有意に識別することができた.
これらの成果は,データ駆動的に決められた細胞の種類を細胞の画像から推定できることを示している.別の視点からは,細胞画像が,データ駆動的に決められた細胞種のマーカーとなりうる,ともいえる.このアプローチは多細胞の成り立ちや各細胞の特性を捉えるために非常に重要である.新しい細胞種や状態を発見した時に,その種や状態を一つ(または数個)の遺伝子の発現量だけで規定できないことはしばしばあるからである.
次に,トランスクリプトームにより規定された細胞グループ(細胞種や状態)に関わらず,個々の細胞において,それぞれの遺伝子の発現量の推定を試みた.まず,先述した3種類の培養細胞を用いたサンプルにおいて,細胞間で遺伝子発現のばらつきが大きい遺伝子を300種類選定し,これらの個々の発現量を細胞画像から推定した.その後,計測した300種類の遺伝子発現量と,推定した300種類の遺伝子発現量を用いて同時に主成分分析を行ったところ,計測値と推定値を用いた双方のプロットの距離は細胞種間の距離より概ね短いことが確認できた(図3).これは,深層学習により,細胞画像から個々の細胞の遺伝子発現状態を推定できる可能性を示唆している(詳細は文献4を参照).
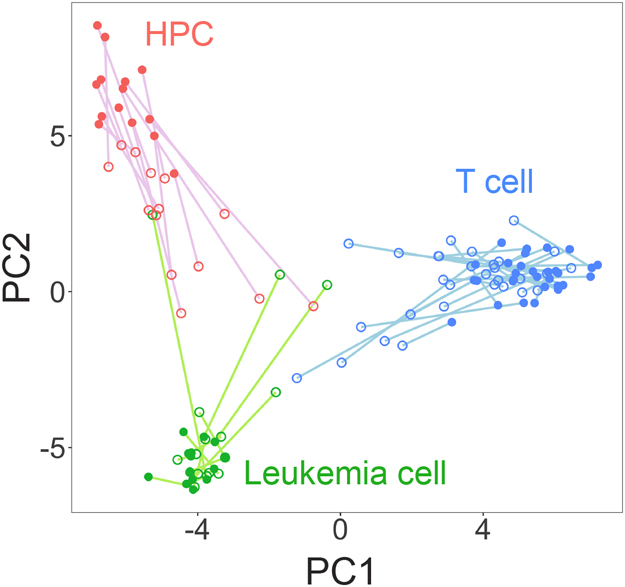
3種類の培養細胞(本文参照)の300遺伝子の発現量を用いた主成分分析.クローズドサークルは各遺伝子の発現量の計測値,オープンサークルは各遺伝子の発現量の推定値を用いた結果.一つの96ウェルプレートから得たデータを示している.文献4から改変.
尚,機械学習では,画像のどのような特徴量を抽出しているのかを知ることは基本的に難しい6).
本研究で示した基盤技術は,いくつかのチャレンジにつながると期待される.
いくつの細胞種を正確に推定できるようになるだろうか.また,何種類の遺伝子の発現量を正確に推定できるだろうか.推定に用いる深層学習のモデルの発展に加えて,画像の種類を増やしたりイメージング手法を改良するなどして,得られる情報を増やすことも重要な要素であろう.
先述したように,1細胞RNA-seqを実施すると,その細胞の未来を知ることは基本的にできない.したがって,例えば,ある段階へ分化する前の細胞の状態を解析することや,結果的に薬剤に耐性があった細胞の薬剤にさらされる前の状態を遺伝子網羅的に調べることなどは難しい.しかしながら,近年,機械学習により,顕微鏡観察から得られた情報を用いて細胞の未来(分化状態)を推定する手法が開発されている7).高い精度で細胞の未来を予測することができれば,ALPSやRNA-seqと組み合わせることで,分化や薬剤耐性などの未来が分かっている(高精度で予測できている)細胞の,分化や薬剤にさらされる前の網羅的解析を実施することが可能であろう.
本研究の成果は,1細胞の遺伝子発現の時間変化を細胞画像から推定できる可能性も示した.精度よく推定できれば,個々の細胞において,データ駆動的に規定される状態や状態間遷移における時間情報(ある状態に滞在する時間はどれくらいか,一方向に状態が変化するだけでなく元に戻ることがあるのか,など)を問うことができる.これは,網羅的解析に時間軸を加えることを意味しており,実現すれば,1細胞研究における重要なツールとなる.
ALPSを開発する際に,敢えて顕微鏡本体は何も改良せず,また,マイクロ流路なども用いないようにした.このため,例えば超解像顕微鏡やシート照明顕微鏡などの様々な顕微鏡システムや計測法に制限を加えずに,ALPSを導入できると考えている.
ALPSによる細胞分取後の解析はRNA-seqだけに制限されない.例えば,ゲノミクスやプロテオミクス,さらにはオミクスではない解析も実施可能である.また,ALPSは細胞などの精製にも利用できる.特に少数の細胞を扱う時に便利であり,例えば100細胞の中から一つのターゲット細胞を取り出したい時などに有効である.ALPSでは大きなサンプルにも対応することができ,300 μm以上のオルガノイドやビーズを分取することもできている.
ALPSを用いたいくつかの共同研究も実施されており,細胞観察と網羅的遺伝子発現解析を組み合わせる手法を用いた幹細胞に関する共同研究も報告されている8).
このような発展性をもつALPSは,1細胞研究だけでなく,様々な生物学研究や医科学研究に貢献できると考えられる.