2025 年 65 巻 5 号 p. 262-265
2025 年 65 巻 5 号 p. 262-265
特定の物をつかむ,というのは,人の基本的な運動の1つであり,UFOキャッチャーで遊ばれたことがある読者も多いであろう.筆者らは,分子レベルでその機能を実装するための第一歩となる実験を行った.今後,分子ロボットへと発展していくことを期待したい.

生活の中において何か目的の物だけをつかむという行動に目を向けたい.例えばゲームセンターのUFOキャッチャーで自分の好きなおもちゃを取るという場面を思い浮かべて頂きたい.これは大量にあるおもちゃの外観から自分の目当ての物を「識別」し,次にそのおもちゃが収まるくらいにUFOキャッチャーの手を広げ「つかむ」という2つの機能から成り立っている.
大量の対象物の中から特定の物だけを取るという普段当たり前の機能も分子レベルで再現することは容易ではない.「識別」機能と対象物のサイズに合った手,つまり「つかむ」ためのキャッチャー構造を作ることが難しいからだ.逆にこういった「識別」と「つかむ」の両機能を分子レベルでも発現できれば,例えば多種多様な性質を持つ試料の中から目的の有用物質を選択的に取得するというようなことができ,医療を始めとする様々な先端分野への応用につながりそうである.こういったいわば分子ロボットとしての機能創成は長年,筆者らが興味を抱いてきた分野でもある.
このような背景のもと,本稿では筆者らが取り組んできた分子ロボットキャッチャーの構築に関する研究を紹介する.具体的には,キャッチャー構造の構築においてはDNA Origami技術を駆使し,捕捉対象物としては近年先端医療分野で注目を集めているEVs(細胞外小胞)に着目した.多種多様な性質,サイズを持つEVsから特定の性質とサイズを持つEVs分画をワンステップで分子ロボットキャッチャーにより捕捉するということを目指したのである1).
EVs(Extra Cellular Vesicles)は脂質二重膜を有する細胞外小胞体の総称でありほとんどの哺乳細胞から分泌されている2).エンドサイトーシスの他,細胞膜の出芽によって形成されるものもある.EVsは分泌された細胞由来のタンパク質を内包,もしくはその細胞由来の抗原をその表面に提示していることも知られている.さらにはmRNA,miRNA等の遺伝情報物質も内包されており,これらが細胞間コミュニケーションに寄与する可能性についても示唆されている3).このようなEVsの一部の分画に関してはエクソソームと呼ばれ,その性質をより詳細に調べることにより次世代の疾患治療や診断等の先端医療につながることが期待されている.
これらの医療応用を実現させる上で一つ重要な「鍵」となるのがEVsの分離手法である.現在,EVsの分離における最も一般的な手法としては密度勾配超遠心を用いたサイズに基づく分離方法が挙げられる.その他にも磁気ビーズ上に抗体を結合させターゲットとなるEVs分画の表面マーカー依存的に分離する手法や,イオン交換担体により電荷依存的にエクソソームを分離する手法も多く提案,開発されている4).さらにはこれらの手法を組み合わせることでより細かくEVsのサブポピュレーションを絞り込み,分離することも行われている.
しかし,多段階の分離では目的分画のEVsを得るための収率は低く,比較的大容量のEVsサンプルが必要になるという欠点がある.このような背景のもと,筆者らはEVsを分類する上で最も重要な指標と考えられるサイズとその表面マーカー,両方の指標で同時に,しかもより少ない試料から目的分画のEVsを分離できるシステムの開発にチャレンジした.
近年,DNAは遺伝伝達物質のみならず超分子構造体を作製するための材料としても注目を浴びている.DNA二重らせん構造の持つ規則性を利用して構造体を作るという試みはDNA Structural Nanotechnologyとして1980年代初頭にSeemanらによってはじめて提唱された5).
その後,本技術は多くの研究を経て2006年にPaul RothemundによってDNA Origami技術と呼ばれる任意の構造体を構築する技術として発展を遂げた6).この技術は長鎖環状DNAに対して相補的なシークエンスを持つ多数の短鎖DNAをハイブリダイゼーションさせることで目的の構造体へと折りたたむというものである.現在では専用ソフトウエアを使うことにより比較的容易に2次元のみならず,3次元構造体7)も設計,作成することができる.
このようなDNA構造体の利点の一つはその構造上の任意の位置にnmオーダーの精度で機能性物質を導入できることにもある.ナノ粒子,タンパク質,アプタマー等の機能性物質をDNA構造体上に導入しそれに応じた特徴的な機能を発現させた報告もされている.特に特定の形状を持った3次元DNA Origami構造体はウィルスや細胞捕捉への応用が報告されている8)が,特定のサイズと分子マーカーを持つ標的だけを捕捉した報告はほとんどなかった.
対象のEVsを捕捉するキャッチャー構造としては筆者らが過去にその構築方法を確立しているDNA Origami技術によるTripod構造を採用した9).Tripod構造が形成する開口内部にエクソソーム捕捉用バインダーを導入し,これにより適切なサイズ=開口部の大きさに合ったエクソソームのみを捕捉するというコンセプトである(図1a).
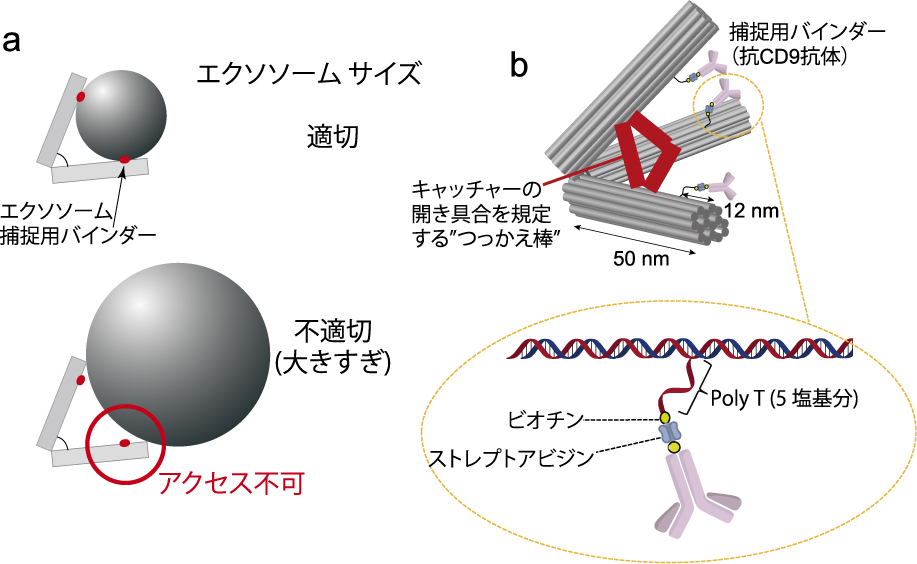
DNA Origami Tripodによるエクソソーム捕捉コンセプトとその設計.a)Tripodの開口部の大きさに合致するエクソソームのみが選択的に捕捉される.b)DNA Origami Tripodの設計詳細.文献1を改変.
Tripod設計の詳細は以下の通りである(図1b).Tripodを形成する3本のアームは剛直性,形状安定性を保つため二重鎖DNAがクロスオーバー(xo)と呼ばれる結束点を介して16本結束された構造となっている.またそれぞれのアーム間には2本の二重鎖DNAが導入されており,これがいわゆるつっかえ棒としてアーム間の角度を形成している.このつっかえ棒の長さを変えることでアーム間の角度を変え,対象となるエクソソームの大きさに合った開口部を形成する.今回の研究においては60°,及び100°の角度を形成するTripodを作成している.
また開口内部の各アーム上にはエクソソームを捕捉するバインダーが導入されている.今回はエクソソーム表面マーカーとして知られるテトラスパニン群の一つである抗CD-9抗体を使用している.60°のDNA Tripodの場合,Tripodもエクソソームも形状的な揺らぎのない剛直な構造体と仮定すると,本構造特性上,50 nm程度のエクソソームがこのDNA Tripodの開口部にピッタリとはまり捕捉される設計になっている.
DNA Origami Tripodに関してはTEMによりその構造形成が確認できる.今回作成した60°及び100°の角度を形成する2種類のTripodもTEM画像からいずれも設計の角度が形成されていることが確認できる(図2a).

DNA Origami Tripod及び捕捉されたエクソソームのTEM像と捕捉エクソソームの粒子径分布.a)60°,100°のDNA Origami TripodのTEM像.スケールバー100 nm.b)60°,100°のDNA Origami Tripodで捕捉したエクソソームのTEM像.c)HT-29細胞から分泌された元々のエクソソーム,DNA Tripodで捕捉されたエクソソーム,磁性粒子で捕捉されたエクソソームの粒子径分布.文献1を改変.
DNA Origami Tripodとヒト大腸腺癌細胞株であるHT-29由来のエクソソームサンプルを混合しインキュベートしたサンプルをTEMで観察するとエクソソームが実際にキャッチャーにより捕捉されている様子が観察される.60°及び100°のDNA Tripodともにそれぞれその開口部より小さいサイズのエクソソームのみを捕捉している(図2b).TEM像からこの捕捉されたエクソソームの粒子径を実際に測定したところ,60°のDNA Tripodにより捕捉されたエクソソームの粒子径分布は47 nm ± 8.7 nmであった.先に述べたように60° DNA Tripodの構造特性から予測される捕捉エクソソームの粒子径は50 nmであるので,概ね予想通りの粒子径となっている.また,HT-29細胞から分泌された元々のエクソソームの粒子径分布は55 ± 21 nm,CD-9抗体を結合させた磁気ビーズにより分離したエクソソームの粒子径分布は60 ± 21 nmとの結果であった(図2c).統計学的な解析を行うと,60°のDNA Tripodで単離したエクソソームは大元のエクソソームの粒子径分布と有意な差があり,サイズ選択的な分離能を発現していることが分かる.
一方で,100°のDNA Tripodでは開口内部に複数のエクソソームが捕捉されてしまっている様子も観察されており,本来の設計よりも小さいサイズのエクソソームが捕捉されている(図2b).本来,本捕捉システムのような複数のバインダーを用いた場合,全てのバインダーに結合した,つまりDNA Tripod開口部の大きさに合致したエクソソームがより選択的に捕捉されることが,筆者らの実施したMonte-Carloシミュレーション上では示されている(図3).しかし,実際には,各バインダーの結合力が強く,3か所結合していないエクソソームでも捕捉されていた.今後はバインダーのアフィニティを低減すること等で捕捉されるエクソソームの粒子径分布が設計通りに変化していくかを確認していく予定である.

Monte-Carloシミュレーションによる捕捉性能予測.a)標的粒子の結合解離を簡易的にシミュレーションした模式図.b)時間が経つと,3箇所で保持されている正しいサイズの標的粒子のみが保持されることが期待される.文献1を改変.
DNA Origami Tripodを用いたサイズ特異的なEVsの溶液中での捕捉が実証できたため,本系を固相上での捕捉システムに展開した.DNA Origami Tripodをエクソソームと混合し12時間後の反応液をガラス基板上に滴下し,DNA Tripodをガラス表面に固定化した.余分なDNA Tripod及びエクソソームを洗い流した後で,全反射蛍光顕微鏡(TIRF)を用いた一分子イメージングによりガラス基板上に固定されたエクソソームの数をDNA Tripodとの共局在で測定した10).ポジティブコントロールの抗CD-9抗体導入系において,共局在率は約15%程度であったのに対して,ネガティブコントロールの抗IgG2a抗体,及び抗体導入なしの系においての共局在化率はそれぞれ7%,4%と,低い結果となった.以上,固相系においてもDNA Tripod によるエクソソームの捕捉が可能であることが明らかになった.
今後,DNA Origamiキャッチャーの立体構造,バインダー導入位置を変えることで生体内のより複雑なタンパク質複合体の捕捉にもチャレンジしたい.また,UFOキャッチャーのアームのように動かすことも可能であり,今後,ナノレベルで様々な物質の検出・捕捉,あるいは処理(つぶしたり,穴をあけたり,くっつけたり)を実現することで,生体内の特定の位置に物質を運び,手術をする分子ロボットの創成もより現実的になっていくと期待される.
本研究は東京大学大学院分子医科学分野の上田卓也教授(研究当時),東京大学分子細胞生物学研究所の泊幸秀教授,上海科技大学のXiaoxia Chen氏,カリフォルニア州立大学サンディエゴ校の増渕岳也氏,他多くの方々にご指導,アドバイスを頂き初めて成し得たものです.この場を借りて厚く御礼申し上げます.
飯沼良介(いいぬま りょうすけ)
JSR株式会社筑波事業所バイオプロセス材料研究チームリーダー(研究・執筆当時)
多田隈尚史(ただくま ひさし)
上海科技大学生命科学技術学院助理教授(PI)