2015 年 14 巻 5 号 p. 155-163
2015 年 14 巻 5 号 p. 155-163
DsRedはDiscosoma種サンゴから単離された赤色蛍光タンパク質(RFP)の一種である.私たちのグループでは,多層フラグメント分子軌道(MFMO)スキームの下,クロモフォアにCIS (D)系の励起状態計算法を用いてDsRedの励起エネルギー・発光エネルギーを算定し,実験のスペクトルの極大値との良好な対応を報告している[Mochizuki et al., Chem. Phys. Lett., 433, 360 (2007) & Taguchi et al., J. Phys. Chem. B, 113, 1153 (2009)].しかし,色素部近傍の水分子,隣接アミノ酸残基の側鎖の配向や構造緩和についての検討は必ずしも明示的になされてはいなかった.そこで今回,励起エネルギーを指標としてこれらの影響を系統的に検証した.その結果,前者では色素部CRQ66や周辺アミノ酸残基と水素結合ネットワークを形成している水2分子の存在,および色素に隣接するSer69の側鎖のOH基の配向,さらに隣接する荷電性のLys163とGlu215の構造の緩和が重要であることが確認された.これらは,結果として既報でのタンパク質構造のモデリングの妥当性を支持するものとなった.
下村らによるオワンクラゲ(Aequorea victoria)からの緑色蛍光タンパク質(GFP) [1]の単離以来,生体工学分野において有用な生体マーカーとして利用されている様々な発色能を有する蛍光タンパク質が多様に開発されてきた.その中の一つが赤色蛍光タンパク質(RFP)の一種で,Discosoma種のサンゴ由来のdrFP583またはDsRedと呼ばれている(以後,DsRedとする) [2,3,4].DsRedの特徴的なスペクトルのエネルギー極大値は励起と発光で,それぞれ558 nm (2.22 eV)と583 nm (2.13 eV)に観測される [2,3].DsRedは,通常は四量体として存在しており,その単量体の構造はβ-barrelと呼ばれるβ-sheetが形成する樽のような形状を成している(Figure 1を参照) [5,6,7,8].DsRedの発色団の重要な色素部位(以下,CRQ66と引用する)は,3つのアミノ酸残基Glu66-Tyr67-Gly68から自己触媒的に形成されるTyr部が脱プロトン化したp-hydroxybenzilidene- imidazolinone部分であり,β-barrel構造のちょうど中心付近に位置している [2,3,4,5,6,7].また,単量体と四量体のエネルギー差は0.01 eVと小さい [8].Figure 2に明示するように,DsRed色素とGFP色素の構造的な差異は酸化ペプチド結合にある.すなわち,GFP色素形成後の酸化反応により,Gln66主鎖のNとCα間に二重結合(acylimine; ―C = N―C = O)が形成され,π共役系が延伸しスペクトルはGFPに比して赤方シフトする.また,DsRedの色素部とPhe65との酸化ペプチド結合はcisとなっていることも特徴的である.Tsienグループ [9,10]は,野生型のDsRedに対して種々の遺伝子改変を行ってmFruitsと呼ばれる一連のRFP群を開発し,生体マーカーとしての応用範囲を大きく広げた.このように,DsRed系の蛍光タンパク質は,実応用面でも高い重要性を持っている [11,12].
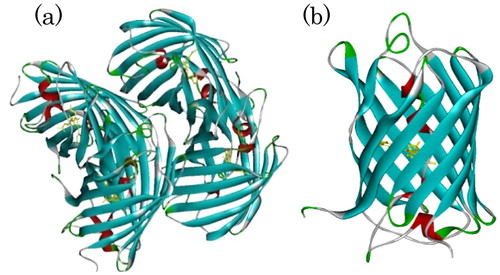
Graphic representation of DsRed (PDB-ID; 1ZGO [7]). (a) tetramer, (b) monomer.

Pigment parts of GFP and DsRed. The π conjugation of DsRed is elongated by an additional oxidation at peptide bond between CRQ66 and Phe65 (marked with dashed line), and thus the optical window is shifted from green to red.
本研究グループでは,フラグメント分子軌道(FMO) [13]スキームの中で量子/古典混成(QM/MM)に発想的には近い多層(MFMO)の扱い [14]に基づき, 1電子励起の配置換相互作用CIS [15]の励起エネルギーに対して2次摂動(MP2)レベルで補正を行うCIS (D) [16]を,系の光応答部に適用するMFMO-CIS (D)法として実装した(その他はHFレベルでの扱い [17,18]).そしてこの計算手法を,DsRedの励起・発光エネルギーを実験値 [2,3]に対して0.1 eVの精度で評価することに成功した [19].続けて,CIS (D) [16]に対して部分的に高次の相関を繰り込んだPR-CIS (Ds)法 [20]を用いた計算では,DsRedの実験値との対応がより向上した [21].文献 [21]では,3種のmFruitsの励起エネルギー [10]も成功裏に算定することが出来た.なお,RFPに対しては本研究グループ以外に多数の理論計算の報告があることを記しておく(タンパク質場を含めたもの [22,23,24,25,26,27,28],色素部に注目したもの [29,30,31,32,33,34]).
さて,タンパク質の理論計算では,用いる構造の妥当性が信頼性の高い結果を得るための重要なファクターとなっていることは言うまでもない.特に,蛍光タンパク質の場合,そのスペクトル特性は,田口らのDsRedとmFruitsの結果 [21]からも示唆されるように色素周辺のタンパク質場の影響にも敏感である.そのため,色素部,隣接アミノ酸残基,ならびに配位水の空間的な配置と構造的な妥当性が信頼性を左右する本質的なファクターとなる.文献 [19,21]では生物物理学的な知見に基づく予備的な検討によって水の個数を決定していたが,系統的な影響を調べることまでは至らなかった.また,色素部CRQに結合したSer69の側鎖の配座についても力場計算で安定と判断される一方の側を選択していた.
こうしたことから,本報ではDsRedの励起エネルギーについて,基本のPDB構造からの再モデリングを行い,色素部に対する水分子の影響,ならびにSer69の配向の影響を系統的に再評価することにした.今回の研究を通じて,励起状態の定量的算定に重要なファクターに関する基礎的な知見を得ることは,水和や立体化学的な制御をより精妙にデザインした新規の光応答タンパク質の開発にとっての一助になることが期待される.
既報のDsRedのMFMO計算 [19,21]では,高解像度1.40 Åの結晶構造(Protein Data Bank [35]のID 1ZGO) [7]が用いられており,本報でも同構造をモデリングの基礎データとした(Figure 1).先ず,四量体から単量体(A chain)を切り出した.発色団の励起に対して決定的な影響を与えている水分子を決めるため,構造中に含める水分子数を色素部CRQ66の重原子から水の重原子の距離を基準に選択し,ぞれぞれ,0分子/2分子(2.8 Å以内)/4分子(3.0 Å以内)/9分子(4.0 Å以内)の計4構造を作製した.なお,どの水分子も構造の揺らぎを評価するパラメーターである温度因子は,結晶水として取り扱ってもよいオーダー(50以下)であった.特に,最近接(2.8 Å以内)の水2分子はCRQ66の共役部位のカルボニル酸素と水素結合を形成している.これら2分子は,Figure 3にスティックモデルで示すように前の計算 [19,21]でも保持されていた.この2つ以外(2.8∼4.0 Å以内)の水7分子(Figure 3にラインモデルで表示)では,CRQ66の共役部位と水素結合を形成しているものはなかったが,CRQ66とSer69の間のペプチド結合に関与しているカルボニル酸素と水素結合を形成しているもの(3.0 Å以内の2分子)や,B構造(後述)にてSer69側鎖のヒドロキシ(OH)基と形成しているものがあった.結局,水分子の数としては,全て無視して0個とした場合,2.8 Å以内(最近接)の2個を含めた場合,3.0 Å以内の4個を含めた場合,Figure 3に示した合計9個を含めた場合の4通りを考えた.これら4つの構造それぞれについて,分子シミュレーションソフトMOE [36]を用いてモデリングを行った.

Conformational differences of Ser69 (marked with dashed line) in the structure including nine water molecules (see text). The two water molecules nearest from CRQ66 (as the DsRed pigment in PDB-ID: 1ZGO [7]) are visible with stick model. The remaining seven molecules with outer positions are represented by the line model. The labels A and B specify the conformation of Ser69 in 1ZGO, and B1 and B2 differ from each other by the orientation of the OH group in the side chain.
結晶構造には占有率の異なるコンフォーマーが含まれていることがあり,1ZGO [7]のA chianでは{Thr21,Phe65,CRQ66,Ser69}の4残基についてそれが見られた.Phe65とCRQ66に関しては,全ての構造においてDsRed色素部の特徴であるcis-ペプチド結合となるようなコンフォーマーを採択した(Figure 2).以下,Ser69の側鎖のOH基の配向が異なる2種類のコンフォーマーでは,占有率0.61の配向を構造A,0.39の配向を構造Bと区別し,配向が発色団の励起に与える影響を検討するために,構造AとB構造を作製した(四量体の残り3つのchainの中で2つはBが優勢であり,配向のデリケートさが伺える).なお,Thr21は発色団領域から十分離れた位置にあるため,占有率の高いコンフォーマー(占有率0.65)を採択した.
次に,MOE [36]のProtonate 3D機能を用いてpH 7.0の条件で水素を付加し,その付加した水素原子のみを古典力場(Amber12: EHT)にて構造最適化した.その結果,肝心の色素部CRQの電荷はphenolate的な-1,タンパク質全体では-2となった(前計算 [19,21]では,Hisの扱いの違いから全体の荷電は+6であった).Ser69のB構造については,Figure 3に示すようにOH基に付加した水素原子の向きが異なるさらに2種類の構造を作製した.一方はOH基の水素がPhe65とCRQ66の間の酸化ペプチド結合に関与しているN原子に配向しており(B1構造とする),もう一方は,周辺アミノ酸残基(Asn42)と水素結合を形成している(B2構造とする).水の個数により4種,Ser69の配向{A, B1, B2}の組み合わせによって,最終的には本モデリングから12構造が得られた.
2.2 古典力場による近接アミノ酸残基の構造緩和2.1節で作成した構造のうち最近接の水2分子を含んだB2構造に関しては,MOEを使って発色団周辺のアミノ酸17残基{Phe14, Met18, Asn42, Leu46, Ser62, Pro63, Lys70, Tyr72, Arg95, Tyr120, Ser146, Glu148, Lys163, Tyr181, Ser197, Gln213, Glu215}の側鎖および水分子について古典力場(Amber12: EHT)で構造最適化した(その他の重原子については全てを固定).ここで,最適化したアミノ酸残基は,相互作用解析結果および発色団に対する立体配置により決定した(詳しくは結果3.4節を参照).
2.3 FMO計算2.1および2.2節で作成した各構造についてMFMO法に基づいた励起状態計算を実行した.注目している励起状態が原子価型と期待されることから,前の計算 [19,21]と同じく基底関数は6-31G*とした.また,FMOプログラムはABINIT-MP,GUIソフトウェアはBioStation Viewerを用いた [37,38].まず,BioStation Viewerにて各アミノ酸残基,色素CRQ66,各水分子がそれぞれ1フラグメントとなるように作成した各構造をフラグメント分割した.MFMO法の励起状態計算領域となるフラグメントは,Phe65とCRQ66,それに今回配向の影響を検証するSer69を1フラグメントとなるようにマージした(既報 [19,21]と同じ).励起エネルギーはCIS (D) [16,18],さらに高次の電子相関を部分的に取り入れて補正したPR-CIS (Ds) [20]で算定した.また,フラグメント間の相互作用エネルギー(IFIE)の解析 [38]をFMO2-MP2/6-31G*レベルで行った.
ここではまず,DsRedの励起エネルギーを考える上で本質的な遷移を同定する.最低励起状態,その上の第二励起状態のCISベクトルを特徴付けている主要な分子軌道は,占有軌道が2つ,非占有軌道が1つの計3つであり,これらをFigure 4に示した.占有軌道は,共役平面全体に広がるπ性のHOMO (マージされた色素部のフラグメント内の番号で147)と,共役面の異なるCRQ66-Phe65間の酸化ペプチド結合に関わるn性の軌道(144)である.一方,非占有軌道はCRQ66の五員環から六員環にかかる部位に関するπ*性のLUMO (148)であった.MFMO-CIS計算より得られた最低励起状態は,主に軌道HOMO-3 (144)からLUMO (148)への遷移によって特徴づけられており,その振動子強度(各構造の平均値)は0.5であった.つまり,第一励起状態はnπ*性である.それに対し,第二励起状態はππ*性のHOMO-LUMO遷移(147 → 148)を主要成分とし,振動子強度が1.3と大きな値を持っており,こちらがDsRedの光応答特性を特徴付けていると考えるべきである.この状況は,以前の報告 [19]と同じであり,実際に最低励起状態にもππ*性の若干の寄与が,逆に第二励起状態にもnπ*性が混入している.色素部だけのモデル計算の最低励起状態はππ*性であり,このnπ*性との干渉はタンパク質場の環境効果であると思われる.これ以後,第二励起エネルギーに注目し,水分子の個数とSer69の配向による影響を12個の構造を通じて見ていく.

Graphic representation of three important MOs of the central fragment (consisting of CRQ66, Phe65 and Se69) calculated at the HF/6-31G* level for the B2 structure with two water molecules. The numbering and characters are indicated for the respective MOs. Orbital phases are shown with red and blue colors.
MFMO計算で算出した(第二)励起エネルギーをTable 1にまとめた.電子相関導入に関する信頼性は表のカラムを右に行くにつれて高くなっており,PR-CIS (Ds)がここでのベストの手法である.実のところ,相関の入らないCISでは1 eV以上の誤差があり,CIS (D)で大幅に改善されるものの,高次の寄与を含むPR-CIS (Ds)の方がより好ましい [17].そこで,これからはPR-CIS (Ds)を基準に議論していく(括弧内の値は,最低励起状態に対する励起エネルギーである).
| without PR | with PR | |||||
| Structure | CIS | CIS (D) | CIS (Ds) | CIS (D) | CIS (Ds) a | |
| No water molecule | ||||||
| A | 3.18 | 1.97 | 2.07 | 1.84 | 1.94 | (1.41) |
| B1 | 3.18 | 1.87 | 1.98 | 1.75 | 1.86 | (1.45) |
| B2 | 3.19 | 2.05 | 2.14 | 1.93 | 2.01 | (1.37) |
| 2 water molecules (within 2.8Å) | ||||||
| A | 3.23 | 2.14 | 2.21 | 2.01 | 2.08 | (1.28) |
| B1 | 3.22 | 2.05 | 2.14 | 1.93 | 2.01 | (1.30) |
| B2 | 3.24 | 2.18 | 2.25 | 2.06 | 2.13 | (1.27) |
| 4 water molecules (within 3Å) | ||||||
| A | 3.24 | 2.07 | 2.16 | 1.94 | 2.03 | (1.41) |
| B1 | 3.24 | 2.00 | 2.09 | 1.88 | 1.97 | (1.43) |
| B2 | 3.25 | 2.14 | 2.22 | 2.02 | 2.09 | (1.37) |
| 9 water molecules (within 4Å) | ||||||
| A | 3.25 | 2.10 | 2.18 | 1.98 | 2.05 | (1.41) |
| B1 | 3.24 | 1.98 | 2.07 | 1.85 | 1.94 | (1.48) |
| B2 | 3.26 | 2.15 | 2.22 | 2.02 | 2.09 | (1.41) |
| Exp. | 2.22 | |||||
a The excitation energy in parenthesis corresponds to the lowest excited state of nπ* type.
Ser69の配向の3種類を通じ,水分子を含まない場合には実験値との誤差は0.2 eVほど過小評価となっているが,CRQ66に最近接の水2分子(2.8 Å以内)を導入するとその誤差はほぼ半減される.さらに,4分子(3.0 Å以内)とするとより改善されるが,9分子(4.0 Å以内)に増加した際は増分の効果はほとんど見受けられなかった.従って,色素CRQ66から最近接の水2分子がDsRedの励起に大きく寄与しており,その励起状態の記述には欠かせない水分子であることが明らかとなった.報告 [19,21]でも,これらの2つの水分子は含めており,モデリングの妥当性が再確認されたと言える.
詳しく調べると,最近接の水2分子はCRQ66の共役部のカルボニル酸素と水素結合を形成しており(Figure 3を参照),一方3.0 Åに範囲を広げた際に加わる2分子はCRQ66-Ser69間のペプチド結合のカルボニル酸素と水素結合を形成している.興味深いことに,これら2分子を導入することで,前節で言及したnπ*とππ*の混ざり合いも低減された(CISベクトルの比較).性格としては,第一励起状態はnπ*性が増加し,今注目している第二励起状態はよりππ*性が増加した.その結果として,励起エネルギーの実験値との対応が水分子無しの場合に比べて改善されたことになるが,励起エネルギーのシフトの方向としては,Boyéらの溶媒効果の議論 [39]に符合している.いずれにせよ,DsRed色素部の2つの低い励起状態におけるnπ*遷移とππ*遷移の相互干渉は強く,最近接水分子2個との水素結合の影響を受けて混じり具合が変わることが改めて示された.
3.3 Ser69側鎖の配向Ser69の配向の違いは,4種の水分子数の場合で同様の傾向を示しており,B2,A,B1の順に励起エネルギーが高くなる.最も実験との対応が良いのはB2である.B2におけるSer69側鎖のOH基は,それぞれ周辺アミノ酸残基のAsn42およびTyr120と水素結合を形成している.一方,対応の悪いB1のOH基は,CRQ66の共役に参加している酸化ペプチド結合のN原子に配向しており,発色団における分子内水素結合とみなすことが出来る.この配向の違いが励起エネルギーの変化として直截に現れている.CISベクトルをチェックするとB1構造では,A構造やB2構造に比べてnπ*性とππ*性の混ざり合いが強まる傾向にあり,実験値との対応も悪くなる.結局,Ser69側鎖の配向としてはB2が妥当であり,色素部周りの水素結合がデリケートな励起エネルギーのチューニングに効いていることが分かる.既述のように,四量体の結晶構造データの中ではAとBのコンフォーマー比はchain毎に異なっており,両者の立体エネルギー差の少なさを映すと共に,結晶環境下と生体環境下でコンフォーマーが異なることを示唆する例とも言えるかもしれない.
Ser69側鎖のOH基の配向を裏付ける実験的考察として,Ser69の配向がDsRed特有の酸化ペプチド結合形成に関与しているとする報告 [5]があり,OHの配向はacylimine部位のN原子に依存するとされている.今回の計算結果は,これを支持する結果となっている.
3.4 発色団周辺アミノ酸残基の構造緩和の励起エネルギーに対する影響評価ここまで,水分子と隣接残基の配向の違いがDsRedの励起エネルギーに対してどのような影響を与えているかを検討し,水素結合を通じて最近接の水2分子とSer69側鎖のB2配向が計算値と実験値との対応では好ましいことが証された.しかし,過小評価の誤差としては0.1 eVあり,PR-CIS (Ds)を使った以前の報告 [21]に比べると思わしくはない.タンパク質全体の正味荷電の違いも考えられるが,他の要因を検討する必要もありそうである.そこで,ここまで重原子部分をPDBの構造に固定していた,色素部と相互作用の大きいアミノ酸残基の側鎖に対し,古典力場による構造緩和を許すことにした(水分子は2個でSer69の配向はB2にセット).緩和の対象は,MP2レベルでのIFIEの結果をベースに選定した(2.2節を参照).
Figure 5に色素CRQ66に対する各アミノ酸残基のIFIEを示した.色素CRQ66は-1の電荷をもっているため,Lys70,Arg95,Glu148,Lys163,Glu215といった荷電性アミノ酸残基とは強く相互作用していることが確認できた.尚,DsRed発色団が静電場によって特徴づけられていることは先行計算 [19,21]からも示されている.この他にも発色団と-20 kcal/mol前後の水素結合由来の安定化相互作用をしている残基(Asn42,Ser146,Gln213等),あるいは-5 kcal/mol以下の分散力由来の弱い相互作用をしている残基(Met18,Leu46,Tyr120等)が存在する.そこで,アミノ酸17残基(Phe14, Met18, Asn42, Leu46, Ser62, Pro63, Lys70, Tyr72, Arg95, Tyr120, Ser146, Glu148, Lys163, Tyr181, Ser197, Gln213, Glu215)の側鎖,および最近接の水2分子について構造を緩和させた(詳しい方法は2.2節参照).

IFIE plots for CRQ66 obtained at the FMO-MP2/6-31G*level.
緩和前と緩和後のアミノ酸残基および水分子の構造変化をFigure 6に示した.CRQ66と静電的な相互作用の大きいLys163とGlu215は変化が大きく,緩和前のIFIEと比較すると各々-11.6 kcal/molと-7.6 kcal/molの安定化の増分を得た(Glu215では反発が減少した).

Superimposed structures around the DsRed pigment. The 1ZGO crystal structure is colored with pink, and the force field-optimized structure is shown with cyan. CRQ66 is identified with yellow.
緩和後の構造を用いて計算した励起エネルギーをTable 2に示す.ここでは,比較しやすいようにTable 1で示した最適化前の励起エネルギーも含めている.新たに算出された励起エネルギーは,緩和前と比べると実験値に対して良い一致を示し,遷移の性格でもππ*性が増加していた.この結果は既報 [21]の値と遜色ないものとなり,色素周囲の荷電性アミノ酸の構造が重要であることが示された.既に触れているようにDsRedタンパク質全体の荷電は,今回の-2と前回[19.21]の+6で異なるのだが,最終的な実験値との対応がどちらも良好なのは,色素部環境において近接・直接的な静電相互作用と水素結合が支配的なことを反映しているとも言えるだろう.
| without PR | with PR | ||||||
| Relaxation | CIS | CIS (D) | CIS (Ds) | CIS (D) | CIS (Ds) b | ||
| Without a | 3.24 | 2.18 | 2.25 | 2.06 | 2.13 | (1.27) | |
| With | 3.28 | 2.29 | 2.34 | 2.16 | 2.22 | (1.15) | |
| Exp. | 2.22 | ||||||
a The same values as in Table 1, with the structure setting of two water molecules and Ser69/B2 conformation.
b The excitation energy in parenthesis corresponds to the lowest excited state of nπ* type.
本稿では,代表的なRFPであるDsRedについて,以前の報告 [19,21]の再訪の形で,PDBデータ1ZGOから改めてモデリングを行い,MFMOスキームの下でPR-CIS (Ds)計算により,色素部CRQ66の周囲の環境が励起エネルギー与える影響を系統的に評価した.構造セットは,CRQ66に近い水分子の個数の違い(①とする),CRQ66に繋がったSer69の側鎖の配向の違い(②)から総計12種類を調製し,励起エネルギーの実験値との対応を調べた.水の影響では,CRQ66の共役部のカルボニル酸素と水素結合を形成しており,重要な役割を持つことが確認された.また,Ser69の側鎖OH基がCRQ66と水素結合をするB2配向が好ましいことが確かめられた.水素結合は量子論的な分極や電子非局在化を伴う相互作用であり,注意深いモデリングが必要であることが示されたとも言える.DsRedの光応答はππ*的な遷移で特徴付けられるが,上記の水素結合はnπ*的な遷移の混じり具合も左右することも分かった.
①と②の因子を押さえるだけでは励起エネルギーの実験値 [2,3]に対して0.1 eVほどの誤差が残るため,CRQ66周囲の相互作用の大きいアミノ酸残基の構造緩和を③として考慮した.具体的には,IFIE解析によって選ばれた17残基の側鎖を古典力場によって最適化した(荷電性のLys163とGlu215の構造変化が大きいことは特記される).この最終構造を用いて求めた励起エネルギーは,実験値の2.22 eVを再現した.文献 [19,21]では,これらの3つの因子は「結果的には適切に取り込まれていた」ために,良好な結果が得られていたことが確かめられたとも言える.いずれにせよ,光応答タンパク質の励起状態の扱いは色素部だけでなく,周囲環境の適切なモデリングが重要であることは間違いない.
本研究を行うに当たり,沖山佳生博士(現在,著者の一人の渡邉と共に理化学研究所に在籍)に技術的なサポートをして頂きましたことを記して,感謝申し上げます.